au sommaire
Les manipulations décrites sont réalisées à 4°C, sauf indication contraire.
2.5.1. ISOLEMENT DES CELLULES
2.5.1.1. Principe
Les granulocytesgranulocytes neutrophilesneutrophiles sont séparés des lymphocyteslymphocytes et des monocytesmonocytes par un gradient de densité obtenu par une solution d'Histopaque-1077 ® (Sigma). Ceci permet de récupérer les lymphocytes et les monocytes.
Ensuite, le culot composé de globules rougesglobules rouges et de granulocytes neutrophiles est remis en suspension dans une solution de chlorure d'ammonium afin d'effectuer une hémolysehémolyse. Les cellules sont recueillies et lavées après centrifugation.
2.5.1.2. Protocole
Les cellules sont prélevées sur héparinate de lithium à partir de sang de volontaires sains. Le sang est dilué au demi dans un tampon Na2HPO4 (PBS) à 0,1 M et à pH 7,4. Au fond de chaque tube, 10 ml d'Histopaque-1077 (Sigma) sont déposés. Après une centrifugation à 400g pendant 30 minutes à température ambiante, les monocytes et les lymphocytes, se trouvant à l'interface plasma-Histopaque, sont récupérés dans un tube à part et mis à centrifuger à 400g pendant 10 minutes, ensuite ils sont mis dans un tampon Hanks' HEPES et conservés à 4°C avant leur utilisation.
Pour récupérer les polymorphonucléaires, 10 ml de plasma sont ajoutés au premier culot composé des hématies et des granulocytes, la suspension est diluée au dixième grâce à une solution isotonique de chlorure d'ammonium (cf. annexe 1), l'hémolyse dure 12 minutes. Les tubes sont ensuite centrifugés à 400g pendant 10 minutes à 4°C, le surnageant est éliminé et les culots lavés par une solution de Hanks'HEPES (cf. annexe 1). Les cellules sont remises en suspension dans la même solution puis centrifugées dans les mêmes conditions que précédemment. Enfin, le surnageant est prélevé et les cellules remises en suspension dans 20 ml de Hanks'HEPES et conservées à 4°C (Cabanis A. et al., 1994).
Une numération est faite sur cellule de Thoma. La vitalité des cellules est évaluée par un test bleu de Trypan. Pour cela, 0,3ml de la suspension sont mélangés avec 0,1ml d'une solution native de bleu de Trypan et mis à incuber à 37°C pendant 10 minutes. Le pourcentage de cellules ne prenant pas la coloration du bleu de Trypan correspond au pourcentage de vitalité cellulaire. L'isolement des cellules est ainsi réalisé avec un degré de pureté d'au moins 95%, la viabilité est également supérieure à 95%.
2.5.2. EVALUATION DE LA CYTOTOXICITE DE LA MOLECULE A TESTER
2.5.2.1. Principe
La lactatelactate déshydrogénase est une enzyme exclusivement cytoplasmique et relativement stable. L'augmentation de l'activité de cette enzyme dans le surnageant des cellules permet de détecter une altération de la perméabilité membranaire.
Le dosagedosage est fondé sur la réaction suivante :
PyruvatePyruvate + NADH,H+ ---> L-lactate + NAD+
Il consiste en la détermination cinétique de l'activité de la lactate déshydrogénase avec suivi de l'absorbance du NADH à la longueur d'onde de 340 nm.
2.5.2.2. Protocole
La cytotoxicité de la molécule à tester est évaluée par le dosage de l'activité enzymatiqueenzymatique de la lactate déshydrogénase (LDH) du surnageant cellulaire d'une suspension de granulocytes neutrophiles ou de lymphocytes, monocytes (5.106 cellules/ml) incubée à 37°C avec l'inhibiteur à différentes concentrations. Les tubes sont centrifugés 5 minutes à 700g, les surnageant sont placés dans les cuves spectrophotométriques. L'activité est mesurée à l'aide de NADH 0,2 mM et de pyruvate 1,6 mM; la cinétique est réalisée à 30°C et lue au spectrophotomètre à 340 nm. L'activité maximale de la LDH libérée dans le surnageant est dosée après la lyse, par choc osmotique, des cellules par ajout de 500 µl d'eau distilléeeau distillée (Levert H. et al., 1998).
Le pourcentage de lactate déshydrogénase relarguée est calculé à partir de l'activité maximale de cette enzyme :
activité LDH totale
X=(activité LDH surnageant de référence)+(activité LDH du lysat)
activité LDH avec inhibiteur (I)
Y=(activité LDH du surnageant avec I)-(activité LDH du surnageant de référence)
%activité LDH=(Y/X)x100
2.5.3. WESTERN BLOT
2.5.3.1. Principe
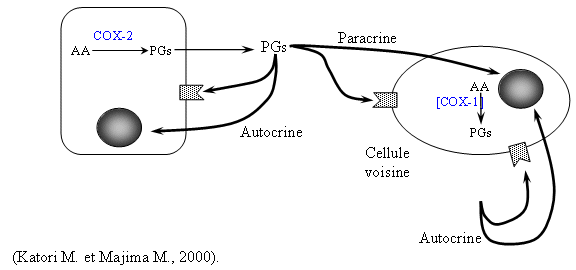
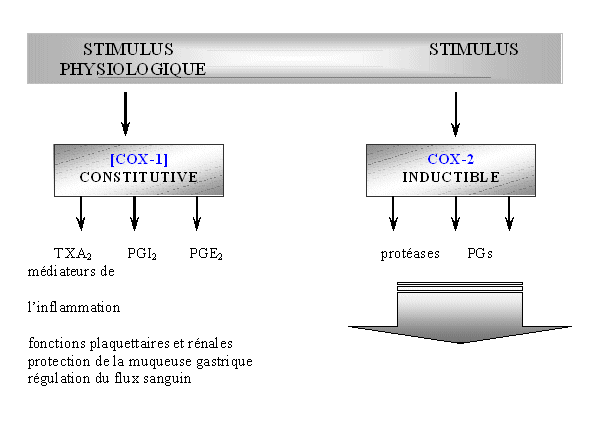
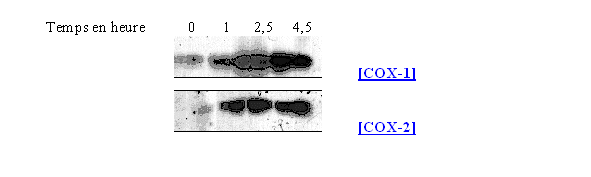
Nous utilisons la technique de western blotwestern blot afin de caractériser la présence des COX-1 et COX-2 après traitement des cellules dans différentes situations.
Les cellules sont incubées avec divers stimulants ou inhibiteurs avec des temps d'activation pouvant varier selon le but recherché. Ces différentes conditions ayant évolué au cours des mises au point, elles seront décrites au niveau des résultats.
Dans tous les cas, les cellules sont lysées, puis les protéinesprotéines sont dénaturées à 100°C en présence de SDS. Ceci entraîne la suppression des structures secondaires, tertiaires et quaternairesquaternaires des protéines permettant ainsi une fixation du SDS sur ces protéines et donc de les charger négativement (transformation en anionsanions).
La migration électrophorétique ne se fera donc plus qu'en fonction de la massemasse moléculaire de chaque protéine, étant donné que chacune possède maintenant la même charge. L'électrophorèseélectrophorèse a pour but de séparer les protéines sur un gelgel de polyacrylamide, ensuite ce gel est mis en contact avec une membrane de nitrocellulose et un courant allant du gel vers la membrane ce qui permet de fixer les protéines contenues dans le gel sur cette membrane.
Le but ensuite est de pouvoir repérer les protéines étudiées sur le support. Pour cela, la membrane est incubée avec un anticorpsanticorps primaire capable de reconnaître la protéine recherchée et de la fixer, puis un anticorps secondaire marqué à la peroxydase est ajouté au milieu afin qu'il puisse se fixer sur le premier anticorps.
La membrane est alors incubée avec du luminolluminol qui est dégradé par la peroxydase en libérant de l'énergieénergie lumineuse capable d'impressionner un film photographique hypersensible. Les protéines apparaissent alors sous la forme de bandes sombres sur le film.
2.5.3.2. Protocole
Les cellules sont dans du tampon Hanks'HEPES à la concentration de 5.106 cellules/ml et à raison de 25.106 cellules par tube pour les granulocytes neutrophiles et de 5.106 cellules pour les lymphocytes, monocytes. Deux incubations de 15 minutes chacune sont réalisées avec du PMSF à 57 µM, ceci pour limiter la dégradation des protéines. Ensuite, chaque milieu est mis en incubation avec l'inhibiteur et/ou l'activateur à 37°C durant un temps donné par l'expérience, selon ce que l'on désire mettre en évidence.
Dans tous les cas, l'arrêt de la stimulationstimulation se fait en ajoutant 4 volumesvolumes de Hanks'HEPES glacé. Chaque milieu est mis à centrifuger à 400g pendant 10 minutes à 4°C afin de récupérer le culot cellulaire.
Les cellules sont remises en suspension dans le tampon de lyse pour western blot (cf. annexe 2) à la concentration de 50.106 cellules /ml. Elles sont soniquées 6 fois 5 secondes, en prenant garde de bien refroidir les tubes entre chaque passage. Tous les échantillons sont dilués au tiers par une solution échantillon (cf. annexe 1) et les milieux sont dénaturés pendant 10 minutes dans un bain-marie à 100°C (Brideau C. et al., 1996, Vane J.R. et Botting R.M., 1998).
L'électrophorèse, adaptée pour le système PROTEAN II (Biorad), est réalisée à 20°C sur un gel de polyacrylamide à 10 % en présence de sodiumsodium dodécyl sulfate (SDS-PAGE) (Laemmli U., 1970).
Dans chaque puits, il est déposé l'équivalent de 130 µg de protéines soit 110 µl de la suspension. La migration électrophorétique se fait à 50 V et pour une duréedurée d'environ 15 heures, en fait c'est le front de migration formé par le bleu de bromophénol qui indique le moment où l'électrophorèse doit être arrêtée.
Les protéines contenues dans le gel sont ensuite transférées sur une membrane de nitrocellulose. Pour cela le gel et la membrane sont mis en sandwiches entre deux feuilles de papier Whatman et deux tampons en fibre, puis l'ensemble est traversé par un courant électriquecourant électrique afin que les protéines se fixent sur la membrane. Cette étape se fait à 24 V pendant 3 heures et à température ambiante. La qualité du transfert est vérifiée en colorant la membrane avec une solution de rouge de Ponceau à 1%, ce qui permet de visualiser l'ensemble des protéines présentes sur la membrane de nitrocellulose.
Cette membrane est incubée pendant 1,5 heure avec une solution pour western blot (cf. annexe 1) contenant du lait à la concentration de 10 %, ceci permettra d'éviter des liaisons non spécifiques des anticorps utilisés par la suite. La solution pour western blot est ensuite employée pour laver la membrane 2 fois 10 minutes. L'anticorps primaire polyclonal de lapin (TEBU), correspondant à l'isoforme de la cyclooxygénase recherchée, est dilué 1000 fois dans la solution pour western blot à laquelle il est ajouté du lait concentré à 1%, puis il est mis en contact avec la membrane pendant une nuit à 4°C ou pendant 1,5 heure à température ambiante.
La membrane est ensuite lavée 2 fois 10 minutes avant de mettre l'anticorps secondaire de singe (TEBU) dirigé contre les chaînes lourdes et légères des anticorps primaires et marqué à la peroxydase. Cet anticorps est dilué 1000 fois dans le même tampon que l'anticorps primaire, cette étape se fait pendant 45 minutes à température ambiante. L'excès d'anticorps est éliminé par deux lavages de 10 minutes.
Les protéines sont maintenant marquées spécifiquement et peuvent être révélées par le kit ECL (Enhanced Chemiluminescence, Amersham). La peroxydase marquant l'anticorps secondaire oxyde le luminol en présence de peroxyde d'hydrogèneperoxyde d'hydrogène. Le luminol excité revient à son état initial en émettant une lumièrelumière à 428 nm détectée par un film photographique (Hyperfilm ECL, Amersham). La membrane est mise au contact du luminol pendant une minute, puis avec le film photographique pour une durée de 30 minutes. La révélation du film est arrêtée lorsque les taches sont suffisamment nettes et marquées. Ceci permet de visualiser la présence de zones sombres correspondant à l'isoforme de la cyclooxygénase recherchée.
La membrane est reprise afin d'être utilisée pour détecter une autre isoenzyme. Elle est mise dans une solution de glycineglycine à 0,1 M à 50°C en pratiquant une légère agitation. Plusieurs lavages successifs sont réalisés afin d'éliminer la totalité de la glycine, la suite du protocoleprotocole est la même que précédemment, à partir de l'étape de saturation de la membrane.
2.5.4. PRODUCTION DU THROMBOXANE B2 PAR COX-1
2.5.4.1. Principe
Les cellules sont incubées en présence de calciumcalcium ionophore afin de permettre la stimulation de la PLAPLA2 et la production de l'acideacide arachidonique contenu dans les phospholipidesphospholipides membranaires. Le maximum de production de thromboxane B2 est obtenu au bout de 1 heure d'activation. Ensuite, les cellules sont centrifugées, le métabolitemétabolite peut alors être extrait du surnageant afin d'être dosé par RIARIA.
La mise en place et l'évolution de ce protocole seront explicitées dans la partie relative aux résultats.
2.5.4.2. Protocole
Les lymphocytes et monocytes à la concentration de 1.106 cellules/ml dans 1 ml sont préincubés pendant 20 minutes à 37°C avec l'inhibiteur potentiel à différentes concentrations.
L'activation des cellules se fait par ajout de CaIo à 50 µM et d'acide arachidonique à 50 µM pendant 1 heure à 37°C. Les milieux sont, ensuite, centrifugés à 400g pendant 10 minutes à 4°C, les surnageants sont mis de coté en vue du dosage de thromboxane B2 par RIA.
2.5.5. PRODUCTION DU THROMBOXANE B2 PAR COX-2
2.5.5.1. Protocole
Les monocytes et lymphocytes sont tout d'abord incubés avec de l'acide acétylsalicylique pour bloquer de façon covalente le site actifsite actif de COX-1, les cellules sont ensuite lavées pour éliminer l'acide acétylsalycilique présent, puis elles sont activées par le LPSLPS dans le but de permettre la transcriptiontranscription et la traduction de COX-2 et ainsi la production de thromboxane B2 dont le maximum est obtenu au bout de 3 heures.
Enfin, les cellules sont centrifugées, le métabolite peut alors être extrait du surnageant afin d'être dosé par RIA.
La mise en place et l'évolution de ce protocole seront explicitées dans la partie relative aux résultats.
2.5.5.2. Protocole
Les lymphocytes et monocytes à 1.106 cellules/ml dans 1 ml sont mis en présence d'acide acétylsalicylique à 500 µM pendant 30 minutes à 37°C. Cette étape est suivie de trois lavages par ajout de 4 ml de Hanks'HEPES puis par une centrifugation de 10 minutes à 400g. Les cellules sont ensuite incubées en présence de la molécule à tester à différentes concentrations pendant 20 minutes à 37°C.
L'activation des cellules se fait par ajout de LPS d'E. coliE. coli à 8 µg/ml et d'acide arachidonique à 50 µM pendant 3 heures à 37°C. Les milieux sont, ensuite, centrifugés à 400g pendant 10 minutes à 4°C, les surnageants sont mis de côté en vue du dosage de thromboxane B2 par RIA.
2.5.6. SOLUBILISATION DES COMPOSES ORIGINAUX
2.5.6.1. Principe
Les molécules originales sont insolubles dans l'eau, dans le tampon (Hanks'HEPES) utilisé pour l'incubation des cellules et dans les solvantssolvants organiques compatibles avec une non toxicitétoxicité vis à vis des monocytes, lymphocytes. Les composés sont tout d'abord dissout dans une substance solubilisante : le polyoxyéthylène sorbitan monolaurate ou polysorbate 20, puis cette solution est diluée par ajout de Hanks'HEPES pour arriver aux concentrations désirées.
2.5.6.2. Protocole
Pour une masse de 1 mg de composés, il est ajouté 500 µl de polysorbate 20. Cette addition se fait en incorporant le solubilisant goutte à goutte et en mélangeant énergiquement afin de dissoudre le maximum de produit dans un minimum de polysorbate 20.
Une première dilution est effectuée en mettant le Hanks'HEPES également goutte à goutte et de façon à avoir un mélange homogène tout au long de cette dilution. Une deuxième dilution peut ensuite être effectuée afin de mettre les composés aux concentrations désirées (Le Hira A., 1997).
Dans le but de vérifier que ces composés sont parfaitement solubilisés, un spectrespectre en UVUV-visible est réalisé sur une partie de la solution et une autre partie de cette même solution est centrifugée à 1000g pendant 15 minutes et un deuxième spectre est réalisé dans les mêmes conditions. En comparant les deux spectres, il est possible de vérifier l'absence de microprécipités.
2.5.7. EXTRACTION CHIMIQUE EN PHASE LIQUIDE
2.5.7.1. Principe
Le thromboxane se trouve au départ dans un tampon qui ne convient pas pour son dosage en RIA, de plus il est trop dilué. L'objectif est donc de transférer le métabolite d'une phase aqueuse vers une phase organique qui sera facilement évaporable permettant de reprendre le thromboxane B2 dans le tampon adéquat et en même temps de le concentrer.
2.5.7.2. Protocole
A 1 ml d'échantillon, il est ajouté 2 ml d'acétoneacétone puis les tubes sont agités pendant 2 minutes, avant d'être centrifugés à 400g pendant 10 minutes à 4°C. Les surnageants sont récupérés et il y est ajouté 2 ml d'hexane, les tubes sont à nouveau agités pendant 2 minutes, puis ils sont centrifugés pendant 5 minutes à 400g à 4°C. Les phases inférieures sont récupérées et amenées à pH 4-4,5 par l'acide citriqueacide citrique. Nous ajoutons ensuite 2 ml de chloroformechloroforme et les tubes sont traités comme précédemment, les phases inférieures sont mises de côté, les phases supérieures sont additionnées de 2 ml de chloroforme et subissent la même opération qu'auparavant. Les nouvelles phases inférieures sont mélangées avec la première (Salmon J.A. et Flower R.J., 1986).
Le chloroforme est évaporé sous courant d'azoteazote, les tubes sont prêts pour le dosage en RIA. Le rendement d'extraction est supérieur à 90%.
2.5.8. DOSAGE DU THROMBOXANE B2 PAR RIA
2.5.8.1. Principe
Ce dosage se fait grâce à un kit RIA (Radio Immuno Assay, Amersham) utilisant de l'iodeiode 125.
Il est basé sur la compétition entre du thromboxane B2 non marqué présent dans le milieu et une quantité fixée de thromboxane marqué à l'iode 125. Il est ajouté un anticorps primaire qui a une haute affinité et spécificité pour le thromboxane B2. Avec une quantité fixée d'anticorps et de ligandligand radioactif, la quantité de ligand radioactif lié reconnue par l'anticorps est inversement proportionnelle à la concentration de ligand non marqué additionné.
L'anticorps lié au thromboxane B2 interagit alors avec un anticorps secondaire lié à une bille d'Amerlex-M. La séparationséparation de la fraction contenant l'anticorps lié est effectuée par centrifugation, la mesure de la radioactivitéradioactivité du culot permet de déterminer la quantité de thromboxane B2 liée à cette fraction. La concentration de thromboxane B2 non marqué est alors déterminée par interpolation à partir d'une courbe étalon.
Les réactions croisées avec d'autres prostanoïdes sontfaibles : 0,3% avec PGE2, 0,15 % avec la 6-cétoPGF1α et 2,5 % avec la PGD2 (données fournies par le fabriquant).
2.5.8.2. Principe
Le thromboxane B2 est repris par 600µl de tampon, et 50 µl sont utilisés pour effectuer le dosage. Pour chaque tube, 100 µl de tampon de dosage sont additionnés avec 50 µl d'échantillon, 50 µl de 125I thromboxane B2 et 50 µl d'anticorps primaire. Il s'ensuit une centrifugation à 800g pendant 3 minutes, puis les milieux sont incubés pendant 15 à 18 heures à 4°C. L'Amerlex-M est ajouté aux milieux à raison de 250 µl et laissé en incubation pendant 10 minutes à température ambiante. Les tubes sont centrifugés à 400g pendant 10 minutes, les surnageants sont éliminés, les culots sont séchés et mis à compter en radioactivité.
2.5.9. ETUDES STATISTIQUES
Les résultats ont été soumis à un test statistique utilisant un test non paramétrique (Student), où les valeurs p