Une équipe de chercheurs a entrepris de cataloguer les différents gènes des bactéries qui peuplent notre corps à l’instar du Human Genome Project répertoriant le génome humain. Les premiers résultats révèlent une diversité génétique totalement inattendue, la plupart des bactéries ayant muté lors de leur séjour dans l’organisme.
au sommaire
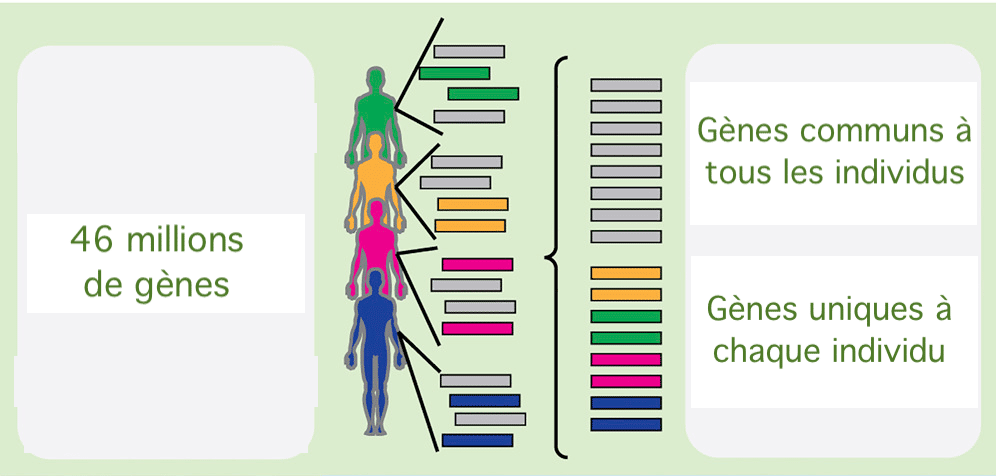
En 2003, le génome humain a été entièrement séquencé grâce au Human Genome Project. On estime ainsi que notre génome contient entre 20.000 et 25.000 gènes codant pour des protéines. Aujourd'hui, les scientifiques s'attaquent à un projet d'une toute autre ampleur : cataloguer les gènes de tous les microbiomes humains. Dans une étude publiée le 14 août dans la revue Cell Host & Microbe, des chercheurs de l'université de Harvard et du Joslin Diabetes Center de Boston dévoilent les premiers résultats issus du séquençage des bactéries et virusvirus de l'intestin et de la bouche de 3.655 individus. Ils ont ainsi décompté pas moins de 45,67 millions de gènes différents, dont la moitié sont uniques à chaque individu (11,8 millions de gènes du microbiome buccalbuccal et 12,6 millions du microbiome intestinal)). En extrapolant à l'ensemble de la population, les chercheurs estiment que le nombre de gènes de l'ensemble des microbiomes humains pourrait être supérieur à celui du nombre d'étoilesétoiles de l'UniversUnivers visible (soit plus d'un quatrillion de gènes ou 1024 étoiles).
Des bactéries qui affectent l’ensemble des fonctions du corps
Le corps humain héberge une quantité considérable de communautés bactériennes dans son intestin, sur sa peau ou dans ses poumonspoumons. On estime que ces bactéries sont 10 fois plus nombreuses que les cellules du corps lui-même. Les dernières avancées scientifiques montrent que ces bactéries jouent un rôle considérable dans le fonctionnement de l’organisme, sur l'immunitéimmunité, les maladies, les allergies, le sommeilsommeil ou même les performances sportives. Or, « de même que deux jumeaux homozygoteshomozygotes ne possèdent pas des gènes strictement identiques, deux bactéries d'une même souche peuvent présenter des différences génétiquesgénétiques », illustre Chirag Pate, de la Harvard Medical School.
Les bactéries mutent pour s’adapter à notre comportement
Mais comment expliquer un telle hétérogénéité ? Les chercheurs ont d'abord pensé que les mutations provenaient de transfert de gène horizontal, un phénomène courant chez les bactéries, où deux cellules proches s'échangent du matériel génétiquematériel génétique. Mais à leur grande surprise, ils ont constaté que moins de 2 % des gènes uniques détectés chez les individus étaient issus de ce processus. L'autre hypothèse envisagée est donc plutôt une évolution adaptative. Lorsque nous changeons de régime alimentaire, que nous prenons tel ou tel médicament, que nous sommes exposés à la pollution ou que nous changeons de stylestyle de vie, les bactéries du microbiome modifient leur ADNADN pour s'adapter à la modification de leur environnement. « C'est grâce à cette diversité génétique qu'un microbemicrobe devient par exemple résistant à un antibiotiqueantibiotique », illustre Braden Tierney, biologiste au Joslin Diabetes Center de Boston et auteur principal de l'étude.
Des traitements probiotiques sur mesure ?
Ces résultats suggèrent que les études portant sur le microbiotemicrobiote doivent être entièrement repensées pour prendre en compte l'hétérogénéité des gènes. Il ne suffira plus de déterminer le type de souche microbienne pour connaître ses effets, il faudra aussi étudier son patrimoine génétique qui est propre à chaque individu, pronostique Alex Kostic, un autre coauteur de l'étude. Bref, élaborer des traitements quasi sur mesure pour chaque personne.
Le projet de recensement des gènes du microbiome humain, intitulé « Univers des gènes microbiens » (The Universe of Microbial Genes), disponible en libre accès sur le site microbial-genes.bio, n'en est qu'à ses débuts.
Séquencer la flore intestinale : un projet ambitieux et prometteur
Article de Destination santé publié le 11/01/2011
Séquencer, cartographier et identifier les gènes des bactéries de notre flore intestinaleflore intestinale : c'est le projet de l'Inra pour étudier les maladies inflammatoires chroniques de l'intestin dont souffrent de nombreux Français. À terme, de nouvelles thérapiesthérapies verront peut-être le jour.
Avec 100.000 milliards de bactéries, voire davantage, la flore intestinale à laquelle le consensus international fait désormais référence sous le nom de microbiote intestinal, livre peu à peu ses secrets. Il existe même aujourd'hui un projet international pour son étude. Baptisé MetaHIT, il a notamment pour objectif de cartographier l'écosystèmeécosystème intestinal. Cela permettrait, dans l'avenir, d'identifier nos prédispositionsprédispositions à certaines maladies. Le point avec OlivierOlivier Goulet, chef du service de gastroentérologie et de nutrition pédiatrique à l'Hôpital Necker-Enfants Malades de Paris.
« La flore intestinale abrite un nombre de bactéries supérieur à celui [contenu dans tout le reste] de notre organisme. Elle porteporte plus de gènes différents que nos propres cellules. Et son poids est supérieur à celui de notre cerveaucerveau. Voilà qui montre bien l'importance de ce monde vivant, hébergé par notre corps, explique Oliver Goulet. Nous interagissons avec lui. Nous pouvons le modifier par notre façon de nous alimenter, de vivre, par notre environnement. »
Lancé en France en 2008 par l'Institut national de la recherche agronomique (Inra), MetaHit (pour Metagenomics of Human Intestinal Tract) est un projet international à part entière. Il est soutenu par l'Union européenne, qui assure plus de la moitié de son financement : 11,4 millions d'euros pour un coût global estimé à 21 millions. Le reste provient du secteur privé. En moins de deux ans, les scientifiques ont identifié plus de 3,3 millions des gènes du microbiote intestinal.
Analyser le microbiote
« Nos recherches visent à en établir la cartographie, mais aussi à identifier ses liens éventuels avec certaines affections. En particulier la maladie de Crohnmaladie de Crohn et l'obésité, précise Oliver Goulet. Nous connaissons désormais les mécanismes à l'origine des maladies inflammatoires chroniques de l'intestin. Et nous savons qu'elles sont en partie provoquées par un déséquilibre de notre flore intestinale. »
Dans un avenir proche et grâce à des puces ADN, il sera possible de mettre au point notre propre profil de flore intestinale. Tout comme nous faisons aujourd'hui notre bilan sanguin. « Nous pourrons donc identifier des prédispositions à certaines maladies. Nous avons devant nous un champ de recherches extraordinaire, qui à terme pourrait ouvrir de nouvelles pistes thérapeutiques. Mais aujourd'hui, nous ne connaissons que la partie émergée de l'iceberg. »
Microbiote ou flore intestinale ? Pourquoi tout à coup, utiliser un mot nouveau pour parler de cette flore qui colonise notre système digestif ? « Le concept de flore intestinale a été utilisé pendant des années car il parle bien au public. Je suis très attaché à cette appellation », nous explique Olivier Goulet. En revanche, « le consensus international de scientifiques préfère s'en tenir au terme de microbiote intestinal : micro au sens microscopique, et bios signifiant vie ».
Source : interview du Pr Olivier Goulet, chef du service de gastroentérologie et de nutrition pédiatrique à l'Hôpital Necker-Enfants Malades (Paris), 5 janvier 2011 ; laboratoire de biologie virtuelle CNRS UMR 6543, faculté des Sciences, Nice-Sophia-Antipolis.
Ce qu’il faut
retenir
- L’univers des gènes microbiens est un projet d’indexation des gènes du microbiome humain.
- La moitié des gènes étant propre à chaque individu, leur nombre pourrait être supérieur à un quatrillion.
- Grâce à ces mutations, les bactéries s’adaptent au comportement de chacun.