au sommaire
On connaît l'opération de GoogleGoogle (Google Image Labeler), qui demande aux internautes de travailler bénévolement en annotant les images de mots clés. Il y a mieux désormais : on peut aider la science en s'amusant. Foldit - littéralement Pliez-la - est une sorte de puzzle tridimensionnel où l'on doit déformer des protéinesprotéines pour trouver des configurations fonctionnelles. On ne gagne rien, si ce n'est une pluie d'étoilesétoiles, mais ce travail est enregistré et pourra contribuer à faire progresser la science.
L'idée de mettre à contribution les propriétaires d'ordinateurs sur ce thème de recherche n'est pas nouvelle. A la manière de Seti@home, le projet Rosetta consiste déjà à faire travailler des micros pour l'étude de la forme des protéines. Mais dans ce cas, seul le processeur se confronte au problème, travaillant en tâche de fond ou quand l'ordinateur n'a rien d'autre à faire. Foldit, lui, réclame la participation active de l'être humain qui doit donner de son temps et de son intelligenceintelligence. En contrepartie, il est censé s'amuser. Les promoteurs du projet, dont David Baker, un biochimistebiochimiste à l'origine de RosettaRosetta, ne proposent rien d'autre qu'un jeu cérébral de plus, mais utile. Plutôt que passer son temps perdu à emplir des grilles de sudoku, pourquoi pas, en effet, exercer ses neuronesneurones à des activités qui pourraient aider la médecine ?
Mais pourquoi les biochimistes ont-ils autant besoin d'assistants ? Parce l'un de leurs sujets de recherche est aujourd'hui de comprendre et même de prédire la fonction des protéines. Ces énormes moléculesmolécules sont formées de centaines voire de milliers d'éléments enchaînés, les acides aminésacides aminés. Leur rôle est fondamental chez tous les êtres vivants. Elles sont pour la plupart des enzymesenzymes, c'est-à-dire des catalyseurscatalyseurs extraordinairement spécifiques et efficaces qui dirigent les réactions chimiquesréactions chimiques dans la cellule ou ailleurs. Dans le sang, c'est une protéine, l'hémoglobinehémoglobine, qui capte et transporte l'oxygène et le gaz carboniquegaz carbonique. Dans les muscles, la myoglobinemyoglobine provoque la contraction des fibres musculairesfibres musculaires.
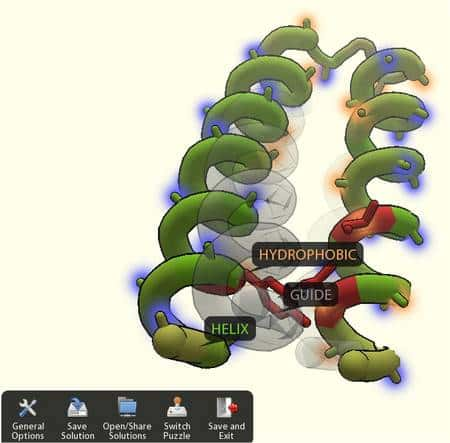
Ici, l'exercice consiste à protéger les sites hydrophobes en les orientant vers l'intérieur de la protéine, entre les deux hélices. (Capture d'écran du logiciel Foldit)
Trouver une forme parmi des milliards
Leur forme est déterminante pour leur action. Par exemple, l'enzyme qui capture le glucoseglucose pour le fixer sur la grosse molécule de glycogèneglycogène, servant de stock de sucresucre pour la cellule, doit posséder un site, c'est-à-dire un assemblage moléculaire, exactement adapté à la forme du glucose, comme une serrure l'est à une clé. Les protéines de ce genre ont souvent deux formes, l'une étant inactive. Le site prévu pour la molécule cible est alors inaccessible. Dans le sang, par exemple, la thrombine (ou thrombase) reste heureusement totalement inactive. Mais qu'une plaie viennent à ouvrir la peau et l'oxygène de l'airair, en venant se fixer sur la thrombine, la rend active. L'enzyme, en se pliant d'une autre manière, libère son site actifsite actif. Elle attrape immédiatement les petites molécules de fibrinogènefibrinogène présentes dans le sang et les agglutinent en longues fibres (la fibrinefibrine), qui s'entremêlent pour former une sorte de gelgel. Et ainsi le sang coagule...
Ce mode de fonctionnement est bien compris mais, pour les milliers de protéines connues, on ignore le plus souvent la ou les formes que prend réellement la molécule dans l'organisme. Car pour chaque protéine, il en existe un très grand nombre. Cette immense chaîne d'acides aminés, hérissée d'appendices pouvant eux-mêmes pivoter sur plusieurs axes, peut en effet se replier de milliards de manières différentes. Parmi toutes les configurations possibles, la protéine optera naturellement pour celles qui correspondent à une énergieénergie minimale. Un logiciel tournant sur un ordinateur, à partir de la composition de la protéine, saura déterminer toutes les formes possibles et calculer celles qui seront stables. Oui mais... il lui faudra des années de calcul pour une seule protéine !
Une démonstration (en anglais) expliquant comment procéder.
Pourtant ce travail est indispensable. On sait déterminer la composition d'une protéine, par analyse chimique ou en séquençant le gènegène qui lui correspond (« qui code pour elle » dirait un biochimiste). Mais pour comprendre à quoi sert cette protéine, il faut en connaître aussi la forme qu'elle adopte dans l'organisme. La compréhension des mécanismes biochimiques, et donc, notamment, la réalisation de médicaments, sont à ce prix.
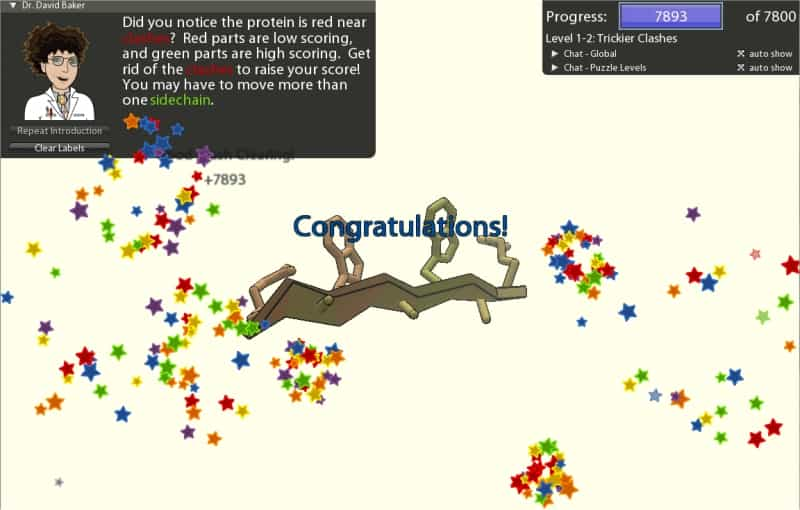
L'idée du projet Foldit est que les logiciels sont moins efficaces qu'un cerveaucerveau humain pour résoudre ce problème, qui s'apparente à un puzzle. L'équipe a donc conçu un véritable jeu, que l'on peut télécharger sur un ordinateur. Le logiciel est massif (53 Mo...) et se connecte à Internet pour récupérer les données à traiter. Pour les premières utilisations, le joueur s'exerce avec des problèmes simples et apprend à utiliser différents outils. La protéine est montrée à l'écran, avec, marquées en rouge, les zones qui posent problème. A la souris, il faut alors faire pivoter des morceaux protéines de sorte d'éloigner des sites trop voisins ou en protéger d'autres en les positionnant vers l'intérieur de la molécule. A condition de ne pas être arrêté par la langue anglaise, on peut facilement se laisser prendre au jeu, qui vaut bien un sudoku ou un mot fléché...