au sommaire
Le Projet génome humain a été lancé en 1990 pour déterminer le séquençage complet de l'ADN humain. Il s'est terminé avec succès en 2003, mais a coûté des centaines de millions de dollars et a pris treize ans. Les chercheurs annoncent que grâce à leurs techniques basées sur un nanopore, ils pourraient très nettement réduire les tarifs et les durées. Pour peut-être, un jour, ne demander que quelques minutes en échange de quelques euros... © Zephyris, Wikipédia, cc by sa 3.0
La génétique peut nous jouer des mauvais tours. Elle est par exemple impliquée dans de nombreuses maladies, comme dans les cancers, le diabète, Alzheimer... La liste est longue. Chacun est donc plus ou moins disposé à présenter des pathologies, et cela peut se lire au niveau de certains gènes. Encore faut-il pouvoir décrypter la séquence nucléotidique qui les compose, de manière à mieux anticiper leur survenue.
Cela ne constitue plus vraiment un obstacle depuis les années 1970, grâce au séquençage de l’ADN, une technique qui permet de constater l'ordre des bases azotées formant la moléculemolécule clé des noyaux cellulaires. Les méthodes ont progressé au fil des décennies, avec des machines à séquencer automatisées, mais cela reste encore coûteux et demande des délais assez longs. À titre indicatif, les appareils les plus performants pourraient décrypter environ 2 millions de nucléotides en une demi-journée... Suffisant donc pour lire quelques gènes. Mais il faut mathématiquement 800 jours (plus de deux ans) à ce type d'appareil pour établir l'ordre des 3,2 milliards de bases qui composent l'ADNADN humain.
Les chercheurs tentent donc d'élaborer des procédés encore plus rapides. Et les pistes explorées pourraient bien améliorer les rendements de lecture. Deux études, publiées dans des revues prestigieuses, proposent de faire passer la molécule dans un trou microscopique de manière à ce que les nucléotides soient obligés de passer un à un, afin de lire à leur passage la signature électrique qu'ils laissent.
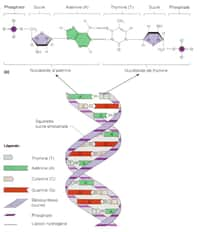
Le séquençage de l’ADN par la porine
Par exemple, les travaux publiés dans Nature Biotechnology, l'œuvre de chercheurs de l'université de Washington (Seattle), font appel à une protéineprotéine et à un moteur moléculaire. En modifiant génétiquement la porine A de la bactériebactérie Mycobacterium segmatis, ils obtiennent une molécule avec un pore d'un nanomètrenanomètre (un milliardième de mètre) de diamètre. Dans cette configuration, l'orifice est si étroit qu'il ne peut laisser passer qu'une seule base d'ADN à la fois.
Ce nanopore est recouvert par une membrane plongée dans une solution de chlorure de potassiumpotassium, dans laquelle on applique une faible tension électrique de manière à créer un déplacement des ionsions à très faible intensité. Cette intensité va alors varier lorsqu'une des quatre bases de l'ADN - adénineadénine, cytosinecytosine, guanineguanine ou thyminethymine - passe au travers du pore, laissant une signature électroniquesignature électronique qui lui est propre.
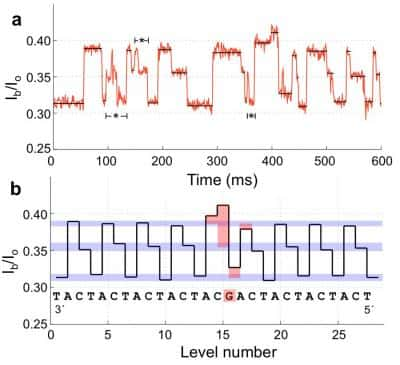
La variation d'intensité en fonction du passage de chaque nucléotide (A, C, T, G) dans le pore est ici nettement visible. Il est donc possible de déterminer assez facilement leur position sur la molécule d'ADN. © University of Washington
Ce principe avait déjà été tenté mais la molécule passait trop vite pour que le détecteur n'ait le temps d'effectuer les mesures précises. De ce fait, ils ont rajouté un moteur moléculaire, une enzymeenzyme appelée ADN polymérasepolymérase phi29, retrouvée dans la bactérie Bacillus subtilisBacillus subtilis contaminée par un virus bactériophagebactériophage. Ce moteur permet de faire passer les nucléotides toutes les 10 ms dans le pore, une vitessevitesse suffisante pour que le capteurcapteur puisse détecter le signal électrique.
Ils ont réussi à lire avec succès six brins d'ADN différents, longs de 42 à 53 nucléotides. Mieux, ils prétendent être en mesure de ne pas se limiter à l'ordre des bases dans la séquence génétique, mais également détecter les modifications épigénétiques, c'est-à-dire la présence ou l'absence de molécules régulatrices de l'expression génique. Or elles interviennent également dans la survenue de certaines pathologies.
Le graphène : récemment découvert, déjà très exploité
Dans l'étude publiée dans Nano Letters par des scientifiques de l'université du Delaware (Newark, États-Unis), il s'agit là encore de faire passer l'acide nucléiqueacide nucléique dans un nanopore, mais cette fois creusé dans une structure en graphènegraphène. Ce composé est issu du graphitegraphite, une macromoléculemacromolécule constituée de couches d'atomesatomes de carbonecarbone, bien connue puisque c'est grâce à elle que les crayons à papier peuvent écrire. Le graphène correspond à un feuillet constitué d'une couche de graphite d'un atome d'épaisseur.
Après plusieurs échecs de différentes équipes ayant utilisé cette technique, les chercheurs ont mis au point des nanorubans avec des motifs très particuliers en zigzags sur les bords. Grâce à un courant électriquecourant électrique qui passe soit verticalement à travers le pore soit transversalement dans la structure de graphène, ils ont pu détecter des changements de conductanceconductance associés au passage de chacune des bases azotées.
Dans les deux cas, les chercheurs estiment que le coût d'utilisation serait inférieur à 1.000 dollars (749 euros), à mettre en regard des centaines de milliers de dollars nécessaires il y a encore quelques années. Et à la vitesse à laquelle les techniques évoluent, ils pensent que le jour où il faudra payer une poignée d'euros pour connaître son génome complet en quelques minutes n'est peut-être pas si loin...