au sommaire

Le séquençage de l'ADN humain est désormais possible, et ce de plus en plus rapidement ! © IRH, Unicef
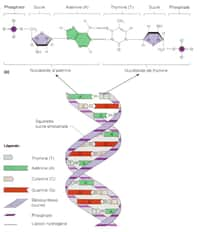
Le séquençage de l’ADN est une technique qui a révolutionné la biologie moléculaire dans les années 1970, soit seulement une vingtaine d'années après l'établissement de la structure de l'ADN par Watson et Crick. L'ADN, le génome contenu dans chacune de nos cellules, contient l'information nécessaire à l'expression des gènes, une information importante aux yeuxyeux des biologistes.
La connaissance de sa séquence, c'est-à-dire la succession des bases de l'ADN (adénine, cytosinecytosine, guanineguanine, thyminethymine), est aujourd'hui de plus en plus facile à déterminer. Futura-Sciences vous donne les clés, sous forme d'un schéma explicatif, pour comprendre le mécanisme du séquençageséquençage de l'ADN.
Décryptage du séquençage de l'ADN
Alors que la méthode de Maxam et Gilbert (la première réalisée) consistait à utiliser les propriétés chimiques des nucléotidesnucléotides, la méthode de Sanger, qui repose sur la biologie moléculaire, est désormais la plus utilisée. Bien que le séquençage ait beaucoup évolué et soit désormais automatisé, il repose généralement sur l'utilisation de composants biologiques qui existent naturellement dans les cellules.
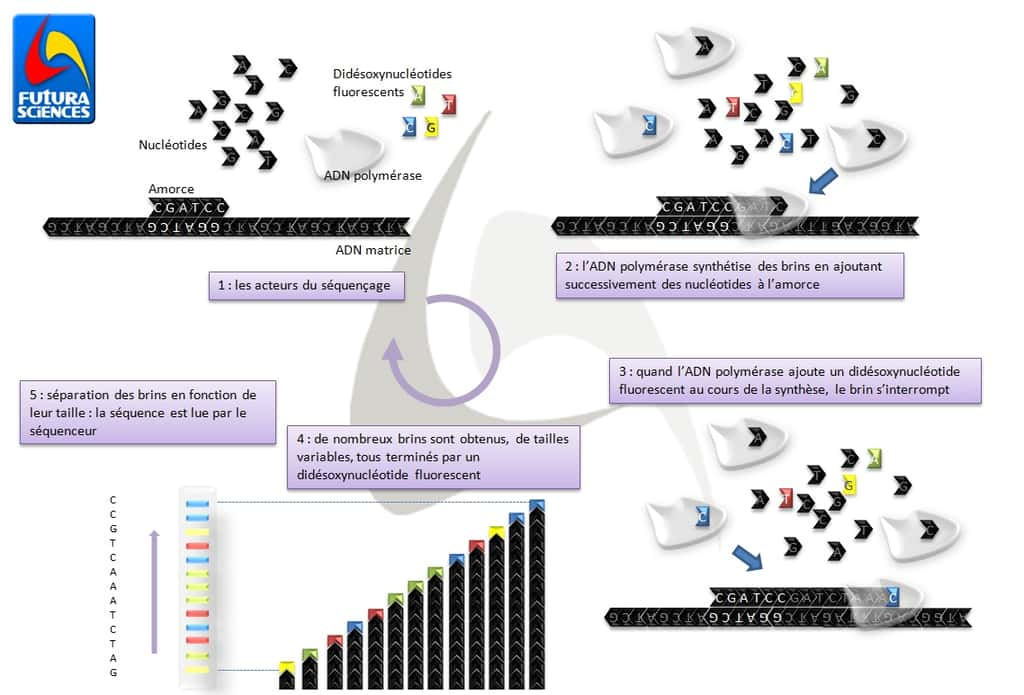
Les différentes étapes du séquençage. © Claire Peltier/Futura-Sciences
- Les différents acteurs du séquençage
- l'ADN provient des organismes dont on souhaite séquencer le génome. L'ADN, le plus souvent sous la forme double-brin, est dénaturé afin de séparer les deux brins. Celui qui sera séquencé s'appelle le brin « matrice » ;
- les nucléotides, ou plutôt désoxynucléotides, sont les briques de l'ADN (A, C, G ou TT). Ils sont attachés les uns aux autres grâce à des liaisons chimiquesliaisons chimiques nécessitant la présence d'un groupement OH particulier (sur le carbonecarbone 3' du riboseribose) ;
- les didésoxynucléotides sont des nucléotides privés du groupement OH en position 3'. Leur incorporation dans une chaîne d'ADN interrompt définitivement la synthèse de l'ADN ;
- une amorce est un brin d'ADN très court (une vingtaine de nucléotide), qui peut s'hybrider à une séquence complémentaire spécifique ;
- l'ADN polymérasepolymérase est une enzymeenzyme qui a pour rôle de copier l'ADN, en synthétisant un brin complémentaire au brin matrice. L'ADN polymérase ne fonctionne qu'en ajoutant des nucléotides à une amorce déjà présente, en fonction de la succession de nucléotides du brin matrice (un A est toujours positionné en face d'un T et un C toujours en face d'un G).
- Les étapes du séquençage
- 1 - Pour le séquençage, tout se passe initialement dans un tube à essaitube à essai, en présence des acteurs de la synthèse de l'ADN : de l'ADN à séquencer, des nucléotides, une amorce et de l'ADN polymérase. Chacun de ces acteurs est présent en grande quantité. La réaction de séquençage fait donc intervenir de multiples réactions.
- 2 - L'ADN polymérase utilise aléatoirement les nucléotides présents dans le milieu pour copier le brin (ou fragment, ou chaîne) matrice en synthétisant un ADN de séquence complémentaire, du moment que la base (A, C, G ou T) est respectée.
- 3 - Lorsque l'ADN polymérase choisit par hasard un didésoxynucléotide (ce qui est rare puisqu'il y en a moins que des nucléotides) et qu'elle l'incorpore dans la chaîne en synthèse, celle-ci s'interrompt prématurément. Puisque chaque didésoxynucléotide est marqué par un fluorochrome différent (A vert, T rouge, G jaune et C bleu), une chaîne qui se termine par exemple par un A sera verte.
- 4 - Puisqu'un grand nombre de réactions de synthèse ont lieu dans le tube, il existe statistiquement des chaînes de toutes les tailles (correspondant à un arrêt de la synthèse à chaque nucléotide) et beaucoup de fragments d'une même taille. Ces chaînes commencent toutes au même endroit sur l'ADN matrice (déterminé par l'amorce utilisée), toutes celles qui possèdent la même longueur se terminent donc par le même didésoxynucléotide marqué.
- 5 - Il est alors possible de séparer les chaînes d'ADN obtenues en fonction de leur taille, sur un gelgel d'acrylamideacrylamide en présence d'un courant électrique. Plus les chaînes sont courtes, plus elles migrent loin et tous les fragments d'une même taille migrent à la même distance. On obtient alors une succession de bandes colorées, chacune correspondant au dernier nucléotide incorporé. Il suffit alors de lire la succession des couleurscouleurs pour connaître l'ordre des nucléotides, c'est-à-dire la séquence de l'ADN, étape assurée automatiquement par les détecteurs du séquenceur.
Historiquement, le marquage était radioactif et l'on ne pouvait donc pas distinguer un didésoxynucléotide des trois autres. Le séquençage était réalisé dans quatre tubes différents, chacun d'entre eux possédant un seul type de didésoxynucléotide. L'analyse des contenus des quatre tubes sur un gel d'acrylamide dans quatre pistes côte à côte montrait alors la succession des bases. Le remplacement du marquage radioactif par des fluorochromes aujourd'hui couramment utilisés, permet de réaliser les quatre réactions dans le même tube. La méthode est limitée au séquençage d'un millier de nucléotides consécutifs, à cause de la faible processivité de l'ADN polymérase qui se détache au cours de la synthèse.
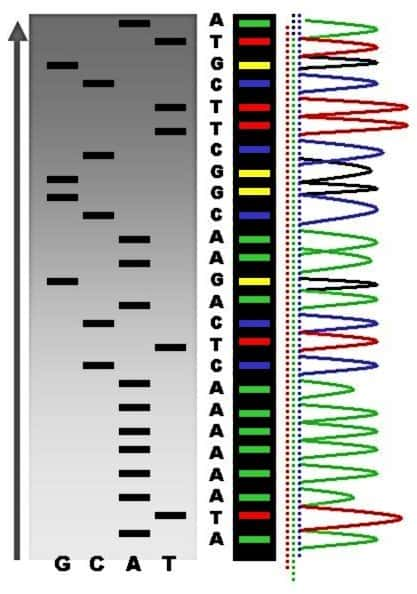
La séquence peut se lire à l'œil sur un gel d'acrylamide grâce aux bandes radioactives visibles dans chaque poche (à gauche, en noir et blanc), ou au séquenceur capable de détecter les intensités de fluorescence de chaque bande (à droite, en couleurs). © Abizar, Wikimedia, CC by-sa 2.5
Évolution des méthodes de séquençage de l'ADN
Pour séquencer un génome, qui comporte des milliers de paires de bases pour les plus petits virusvirus, à quelques milliards pour l'Homme ou certaines plantes, il faut donc pouvoir réaliser de multiples séquençages. Il faut ensuite reconstituer l'ordre des fragments obtenus par recouvrement des séquences. Cela se fait à l'aide de programmes informatiques, mais l'obtention d'un génome dans son intégralité est encore un mythe : on obtient dans le meilleur des cas 95 % du génome.
De nouvelles technologies sont progressivement apparues suite à la demande de plus en plus de laboratoires pour séquencer rapidement et à faibles coûts. C'est ainsi qu'est né le pyroséquençage 454, toujours basé sur le même principe, mais qui utilise des volumesvolumes de réactions très petits (quelques picolitres). D'autres types de séquençages sont également basés sur l'utilisation de désoxynucléotides fluorescents et d'ADN polymérases : Solexa, Solid ou encore Helicos tSMS.
Cette dernière méthode permet de séquencer individuellement chaque moléculemolécule d'ADN. Les brins complémentaires de morceaux d'ADN très courts attachés sur une surface sont tour à tour additionnés d'un seul nucléotide fluorescent. À chaque cycle, la localisation de la fluorescence provenant des brins est enregistrée par des caméras, permettant ainsi de suivre au fil des cycles la séquence de tous les brins.
De nouvelles technologies totalement différentes sont également développées, présentant l'avantage de ne pas avoir à répliquer l'ADN ni d'utiliser de fluorochromes. Sous la forme d'une puce dotée de nanopores, la machine capte directement les signaux électriques de chaque base d'ADN qui traverse le pore et permet de séquencer un petit génome (bactériebactérie) en un temps record et pour un prix dérisoire.
Il y a fort à parier que les évolutions ne s'arrêteront pas en si bon chemin...