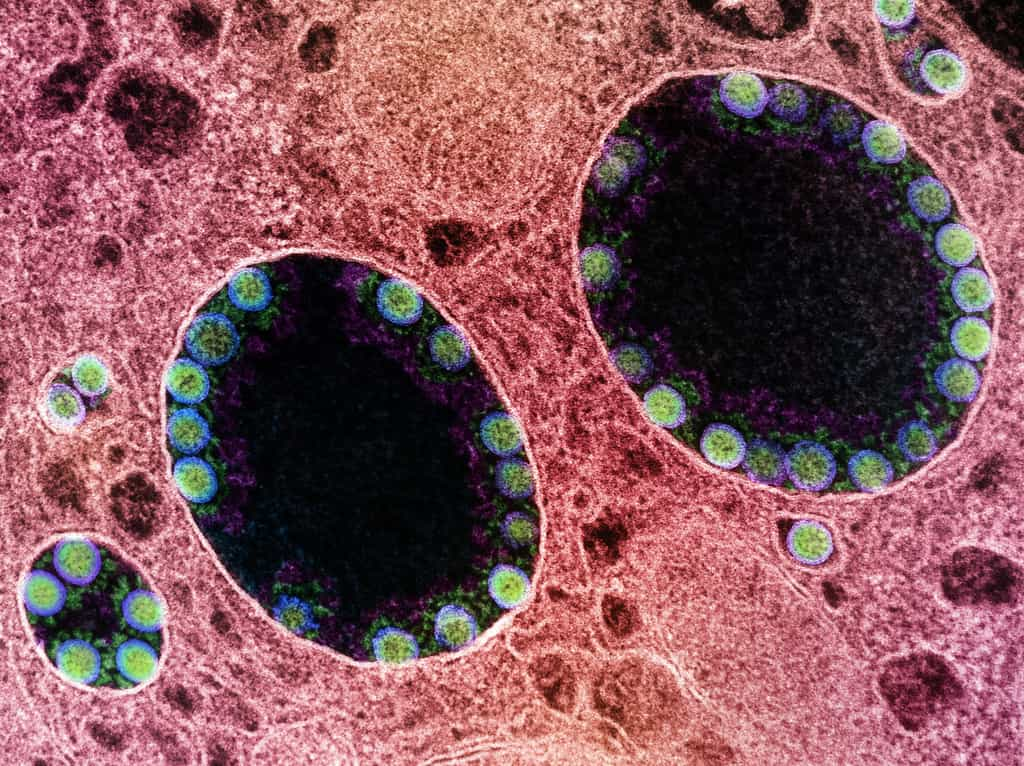
Des chercheurs de l’Institut Pasteur ont isolé des virus de chauves-souris présentant des similitudes extrêmement élevées avec le coronavirus SARS-CoV-2 à l’origine de la pandémie de Covid-19. Ces virus pourraient constituer le chaînon manquant expliquant le passage à l’Homme. Une découverte qui renforce l’hypothèse d’une origine naturelle du SARS-CoV-2 mais qui montre aussi que ce genre de dangereux virus est bien plus répandu dans la nature qu’on ne le pense.
au sommaire
En février 2020, des chercheurs chinois avaient découvert chez des chauves-sourischauves-souris au Yunnan une souche de coronavirus nommée RaTG13 et identique à 96,2 % au SARS-CoV-2, le virus à l'origine de la pandémie actuelle. En novembre, des chercheurs de l'Institut Pasteur en France avaient isolé trois coronavirus chez des chauves-souris au Laos partageant de grandes similitudes avec le SARS-CoV-2 (lire article ci-dessous). Dans une nouvelle analyse livrée à Nature, cette découverte est aujourd'hui confirmée : les trois souches, nommées Banal-52, Banal-103 et Banal-236, partagent à 95 % leur génome avec le SARS-CoV-2, et l'un d'eux, Banal-52, serait même similaire à 96,8 %, soit le plus proche parent du SARS-CoV-2 jamais identifié. « Cela renforce l'hypothèse selon laquelle le SARS-CoV-2 a une origine naturelle [et n'est pas issu d'une manipulation en laboratoire, ndlr], mais fait aussi craindre qu'il existe de nombreux coronavirus dans la nature susceptibles d'infecter l'humain », atteste Nature.
Des virus de chauves-souris capables pour la première fois de se lier à un récepteur humain
Jusqu'à présent, aucun des virus trouvés chez les chauves-souris ne présentait des caractéristiques suffisantes pour expliquer leur transmission à l'humain. Une des clés de cette infection réside notamment dans la séquence codant la protéineprotéine de pointe présente à la surface du SARS-CoV-2. Cette séquence appelée RBD (receptor binding domain)) est essentielle car elle détermine l'affinité de liaison du virus au récepteur ACE2, présent sur les cellules humaines et qui sert de porteporte d'entrée au virus.
Malgré ses similitudes, RaTG13 montre ainsi une faible affinité avec la séquence RBD du SARS-CoV-2, avec seulement 11 acides aminésacides aminés sur 17 identiques. La capacité d'infection de RaTG13 reste donc limitée. « Aucun virus proche du SARS-CoV-2 n'utilise ACE2 pour pénétrer dans les cellules humaines », observent les auteurs de l'article (encore non relu) publié sur Research Square. Dans l'autre sens, le SARS-CoV-2 infecte mal les chauves-souris et les cellules de celles testées jusqu'à présent. Bref, il manque une pièce du puzzle pour expliquer le passage du virus à l'Homme, ce qui a soutenu chez certains l'hypothèse d'une fuite de laboratoire.
Or, les trois virus récoltés parmi des échantillons de salivesalive, de fècesfèces et d'urine de 645 chauves-souris dans des grottes du nord du Laos semblent eux capables de se lier au fameux récepteur ACE2. Le RBD de Banal-52 ne diffère par exemple que d'un seul nucléotidenucléotide par rapport au SARS-CoV-2 et Banal-236 partage 15 des 17 nucleotides de son RBD. Les chercheurs ont également mené des tests en laboratoire qui ont confirmé que Banal-236 parvenait bien à se joindre au récepteur ACE2 et donc à infecter les cellules humaines.
Le mystère de l'arrivée du virus en Chine
Reste néanmoins quelques chaînons manquantschaînons manquants pour expliquer le passage du virus à l'humain. Tous les coronavirus trouvés jusqu’à présent sont dépourvus d'une séquence particulière appelée site de clivageclivage de la furine (présente sur le génome du SARS-CoV-2), qui joue un rôle majeur dans la fusionfusion entre les membranes virale et cellulaire et qui est associée à une pathogénicitépathogénicité accrue chez l'Homme. Autre mystère : l'étude ne précise pas comment ces virus découverts au Laos auraient pu voyager jusqu'à Wuhan, en Chine centrale, là où a débuté la pandémie de Covid-19Covid-19. Une autre étude (non relue elle aussi) a prélevé des échantillons de 13.000 chauves-souris entre 2016 et 2021 dans toute la Chine, et les chercheurs n'ont trouvé aucun proche parent du SARS-CoV-2, concluant donc que ces derniers sont « extrêmement rares » en Chine.
Les grottes chinoises, un écosystème idéal pour l’émergence de dangereux virus
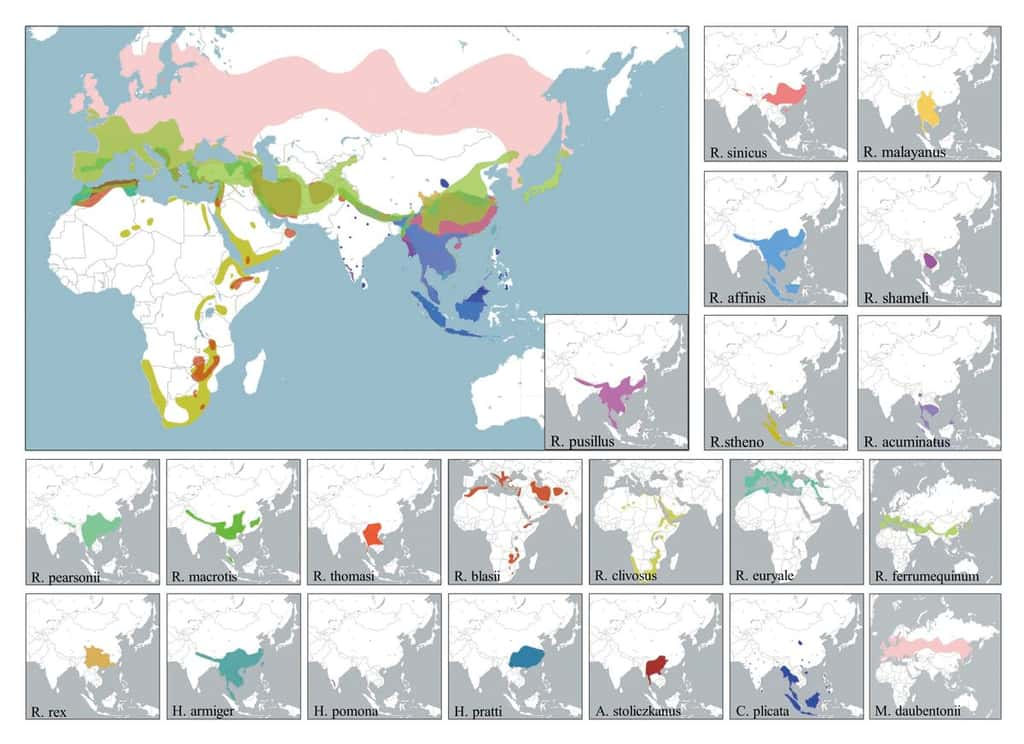
L'hypothèse la plus probable est finalement celle d'une recombinaisonrecombinaison entre plusieurs coronavirus de chauves-souris, dont les séquences génétiquesgénétiques se seraient mélangées pour produire une souche particulièrement dangereuse pour l'Homme. « Le SRASSRAS-CoV-2 pourrait résulter d'une recombinaison des séquences préexistantes chez les chauves-souris Rhinolophus vivant dans les vastes systèmes de grottes calcairescalcaires d'Asie du Sud-Est et de Chine du Sud, qui offrent des conditions idéales pour les interactions inter-espècesespèces », avancent ainsi les chercheurs de l'Institut Pasteur. Un écosystèmeécosystème très spécifique qui expliquerait aussi pourquoi la majorité des épidémiesépidémies démarrent en Asie du Sud-Est.
Des coronavirus proches du SARS-CoV-2 isolés au Cambodge et au Japon
Article de Julie KernJulie Kern publié le 27/11/2020
Des scientifiques au Cambodge et au Japon ont isolé des nouvelles souches de coronavirus d'échantillons de chauves-souris. Sont-ils liés de près ou de loin à la souche pandémique SARS-CoV-2 ?
L'enquête généalogique sur les parents du SARS-CoV-2 et son réservoir naturel connaît un nouveau souffle. Un peu par surprise, plusieurs échantillons, issus de chauves-souris à neznez en feuille (du genre Rhinolophus), conservés dans deux congélateurs de laboratoire, un au Cambodge et l'autre au Japon, ont révélé l'existence de nouvelles souches de coronavirus. Leur lien génétique avec le SARS-CoV-2 est au cœur des attentions.
Dans un congélateur cambodgien
Tout d'abord, regardons rapidement l'arbrearbre généalogique du SARS-CoV-2 tel que nous le connaissons actuellement. Le SARS-CoV-2 est un coronavirus de la famille des Betacoronavirus, il appartient plus précisément au sous-genre Sarbecovirus. On y retrouve les SARS-CoV-1 et 2 et d'autres coronavirus issus des chauves-souris.
Dans les Sarbecovirus, on trouve également le coronavirus RaTG13. Ce dernier est le plus proche parent connu du SARS-CoV-2 actuellement, avec environ 96 % de similitude. RaTG13 est issu d'une espèce de chauve-souris vivant en Chine, Rhinolophus affinis.
Il est difficile de placer la trouvaille des scientifiques cambodgiens dans cet arbre généalogique. Les scientifiques ont communiqué leur découverte de ce nouveau coronavirus à Nature, mais pour le moment sa séquence génétique n'est que partielle et n'a pas été publiée. Ce qui est sûr, c'est qu'il provient aussi d'une chauve-souris, Rhinolophus shameli, capturée au nord du Cambodge en 2010.
S'il s'avère que ce coronavirus est proche du SARS-CoV-2, plus proche même que RaTG13, il pourrait détenir de précieuses informations sur comment le SARS-CoV-2 a fait son chemin entre les chauves-souris et l'être humain. Sinon, il est une preuve supplémentaire que les chauves-souris sont un réservoir important de coronavirus en Chine et dans d'autres pays asiatiques.

Un autre parent du coronavirus SARS-CoV-2 au Japon
Concernant le coronavirus japonais, appelé Rc-o319, on dispose d'un peu plus d'informations. Sa description a été publiée par les scientifiques du National Institute of Infectious Diseases de Tokyo, au début de mois de novembre. Rc-o319 est issu de Rhinolophus cornutus, une petite chauve-souris endémiqueendémique de l'archipelarchipel japonais, dont quatre spécimens ont été capturés en 2013 pour en collecter les fèces.
Les scientifiques ont observé 81,47 % d'identité avec le SARS-CoV-2, ce qui n'est pas suffisant pour en déduire des informations sur l'origine de la pandémie actuelle ou le considérer comme un ancêtre potentiel. Aaron Irving, un chercheur en maladie infectieuse à l'université du Zhejiang à Hangzhou, a indiqué à Nature, qu'il faudrait qu'un coronavirus soit à 99 % similaire au SARS-CoV-2 pour être considéré comme un ancêtre direct.
L'enquête se poursuit donc, mais il est certain que les coronavirus prospèrent dans les chauves-souris du genre Rhinolophus depuis des années et bien avant que le SARS-CoV-2 ne révèle son existence. Le coronavirus responsable de la pandémie de Covid-19 n'est pas apparu du jour au lendemain, et encore moins dans les tiroirs d'un congélateur de laboratoire, il est le fruit d'une longue histoire évolutive qui nous échappe encore.