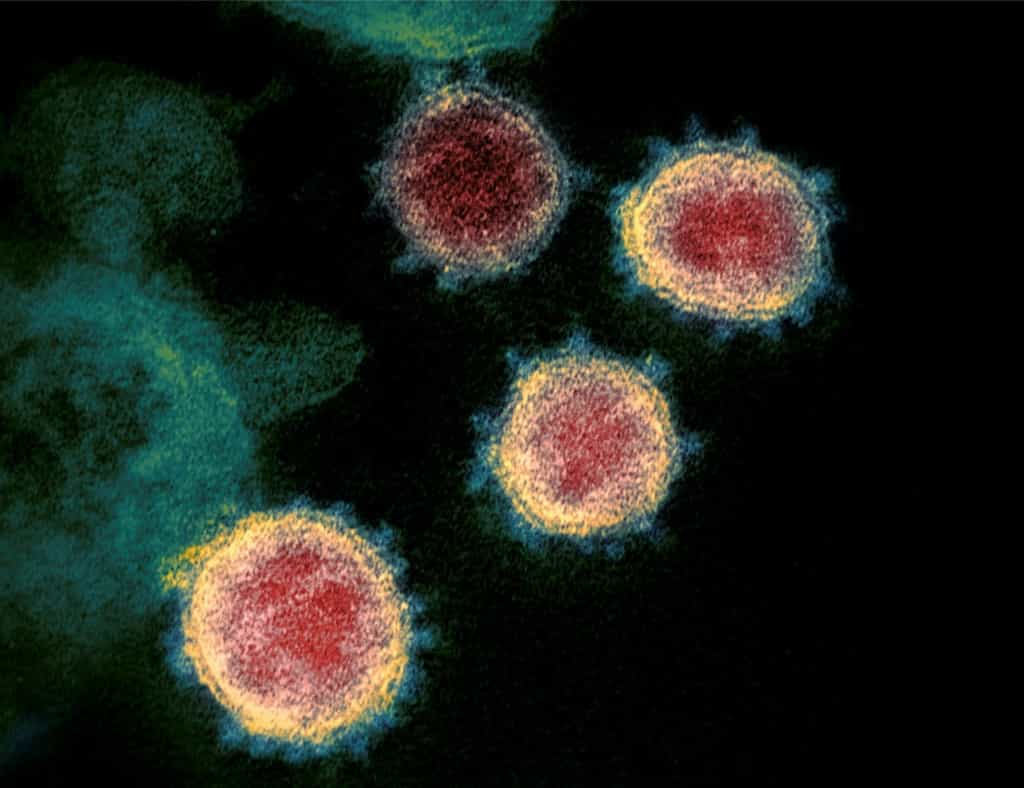
Assez rapidement après l’identification du SARS-CoV-2, les données scientifiques ont permis d’établir que le virus entrait dans les cellules via le récepteur ACE2. Une recherche, qui a rassemblé des laboratoires du monde entier, vient de mettre en lumière les trois populations cellulaires les plus susceptibles d’être infectées.
au sommaire
Grâce à l'étude de la spike protéine qui hérisse le SARS-CoV-2, nous savons que le coronavirus se fixe sur le récepteur ACE2. Une deuxième enzymeenzyme cellulaire, TMPRSS2, participe à l'activation de la spike protéine et autorise alors le virus à entrer dans la cellule-cible.
Une étude, publiée dans la revue Cell et menée par des chercheurs d'Harvard, du MIT mais aussi de l'Inserm et du CNRS en France, a identifié précisément les populations cellulaires susceptibles d'être infectées dans le nez, les poumons et l'intestin.
Les trois populations cellulaires les plus vulnérables
L'étude est basée sur un jeu de données regroupant les ARNARN exprimés par différentes populations cellulaires construites dans différents organes avant l'épidémieépidémie de Covid-19Covid-19. En identifiant les cellules exprimant les ARN issus de la transcriptiontranscription des gènesgènes du récepteur ACE2 et de l'enzyme TMPRSS2, les chercheurs ont mis en lumièrelumière les cellules potentiellement infectables par le coronavirus.
Trois populations cellulaires expriment le plus les ARN des protéines ACE2 et TMPRSS2 :
- les cellules caliciformes des voies aériennes supérieures ;
- les pneumocytespneumocytes de type II qui tapissent les alvéoles pulmonairesalvéoles pulmonaires ;
- les entérocytes de la muqueusemuqueuse intestinale.
L’effet inattendu des interférons sur le récepteur du virus
De plus, au fil de leurs expériences, les chercheurs ont mis le doigt sur un lien inattendu entre ACE2 et l'inflammationinflammation. En effet, l'expression du gène ACE2 est corrélée avec celle de gènes activés par les interféronsinterférons. Pour en avoir le cœur net, les scientifiques ont stimulé des cellules tapissant les voies respiratoires avec de l'interféron. Ce traitement a activé l'expression du gène ACE2.
Les interférons sont des moléculesmolécules de défense produites par les cellules lors d'une infection. Mais le coronavirus semblerait être capable de tourner ce mécanisme de défense à son avantage en entrant via un récepteur surexprimé en présence d'interférons.
« Ce n'est pas le seul exemple connu. Il y a d'autres coronavirus qui ciblent spécifiquement des gènes stimulés par les interférons comme porteporte d'entrée dans les cellules », explique Jose Ordovas-Montanes, le superviseur de cette étude affilié à Harvard, dans un communiqué de presse.
Les interférons sont aussi utilisés comme médicaments anti-infectieux. Par exemple, les hépatites Chépatites C chroniques sont traitées par interférons alpha-2b. Mais, dans le cas du SARS-CoV-2 cela semble plus complexe : les interférons sont efficaces pour lutter contre les infections virales mais ils risqueraient aussi de multiplier les cellules-cibles pour le coronavirus. Comme pour toutes les pistes de traitement aujourd'hui explorées, celle-ci aussi nécessite des recherches poussées et rigoureuses.
Covid-19
Pour en savoir plus sur l'évolution du coronavirus ou Covid-19
Retrouvez tous nos articles sur le coronavirus dans notre catégorie dédiée.
Concernant les actualités de la pandémie :
- Coronavirus : le mystère des patients qui ne guérissent jamais
- Vaccins à ARN messager : quels sont les risques pour notre génome ?
- Quelles sont les différences entre les vaccins Moderna et Pfizer ?
- AstraZeneca : que sait-on des patients victimes de thrombose après l'injection du vaccin ?
- Quels impacts du SARS-CoV-2 sur le cerveau ?
Symptômes du Covid-19 : le diabète et l'hypertension comme facteurs aggravants
Nos questions-réponses :