Un traitement possible contre le Covid-19 suppose de modéliser des protéines impliquées dans la structure du virus. L'opération demande une grande puissance de calcul qui est atteinte en partageant les opérations à mener sur des ordinateurs à l'échelle de la Planète bleue grâce au logiciel Folding@home. Le Cern vient de joindre à ces ordinateurs certaines des machines de son centre de calcul.
au sommaire
Il y a quelque temps, Futura vous avait proposé de vous joindre à la lutte mondiale contre le Covid-19Covid-19 de chez vous en installant le logiciellogiciel Folding@home. Il s'exécute pendant que vous utilisez votre ordinateurordinateur sans que vous le remarquiez et il permet de faire du calcul distribué, lequel consiste à découper en petites unités indépendantes des calculs à effectuer pour les répartir ensuite sur des ordinateurs personnels (MacMac ou PCPC), fonctionnant aussi bien sous Windows que sous macOS ou LinuxLinux.
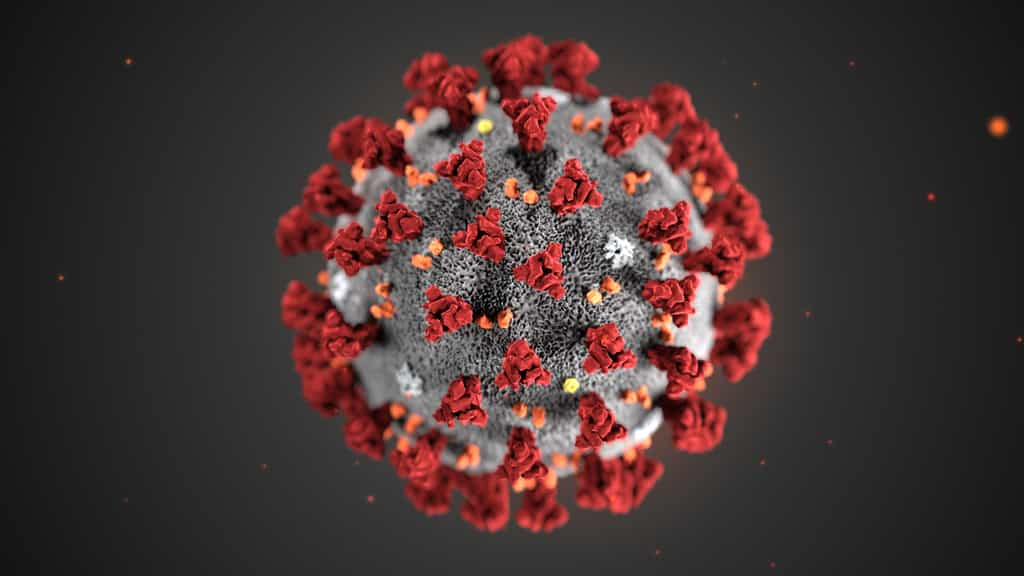
En l'occurrence, les calculs pour lesquels on vous demande de l'aide portent - mais pas seulement car d'autres traitements pour d'autres maladies sont aussi étudiés avec ce logiciel - sur la modélisation de la structure et de la dynamique d’une protéine à la surface du Covid-19 (voir l'illustration créée aux Centers for Disease ControlCenters for Disease Control and Prevention)). Elle intervient dans les dégâts causés par la maladie en se liantliant à une protéineprotéine réceptrice sur une cellule pulmonaire. Un traitement consisterait donc à trouver un anticorpsanticorps thérapeutique, c'est-à-dire un type de protéine qui peut empêcher la protéine virale de se lier à son récepteur, empêchant ainsi le virus d'infecter la cellule pulmonaire. C'est là qu'intervient la modélisation par une technique dite de dynamique moléculaire de la protéine du Covid-19.
Le projet Folding@home fonctionne grâce au réseau mondial d'InternetInternet qui est devenu une illustration frappante de certaines idées visionnaires exposées par le géologuegéologue, paléontologuepaléontologue et philosophe Teilhard de Chardin, avec le concept de Noosphère, et celle d'Arthur Clarke qui anticipait, lui aussi, l'arrivée du fameux village global selon l'expression que l'on doit au philosophe et sociologue des médias canadiens Marshall McLuhan.
Une présentation du Cern. © Cern
Folding@home au lieu de naissance du Web
Or rappelons-le, le Web que nous connaissons est né au Cern il y a 30 ans. Le célèbre laboratoire européen, où l'on a fait la découverte des bosons de Brout-Englert-Higgs, W et Z est lui-même un grand consommateur de calculs lorsqu'il analyse les produits des collisions de protons dans les détecteurs géants du LHC à la recherche d'une nouvelle physique. Ces calculs concernent toute la communauté de la physique des particules mondiale qui travaille sur le LHC. Le Cern avait lui-même suivi la voie de Folding@home en proposant aux internautes de la noosphère de rejoindre la quête de nouvelles particules via le projet de calculs partagés LHC@home. C'est donc sans surprise que l'on vient d'apprendre, par un communiqué du Cern, que le laboratoire avait lui aussi mis à la disposition de Folding@home une partie de la puissance de calcul de ses ordinateurs, environ 10.000 cœurs de processeurs de son principal centre de calcul.
Le Cern précise cependant dans son communiqué que : « Cela ne représente toutefois qu'un tiers environ des "opérations de calcul" que l'équipe du Cern a réalisées pour Folding@home. Le reste correspond à des contributions faites directement par les sites de calcul pour le LHC. Ensemble, à la date du 21 avril, le Cern et les sites de calcul pour le LHC venaient au 87e rang des contributeurs ».
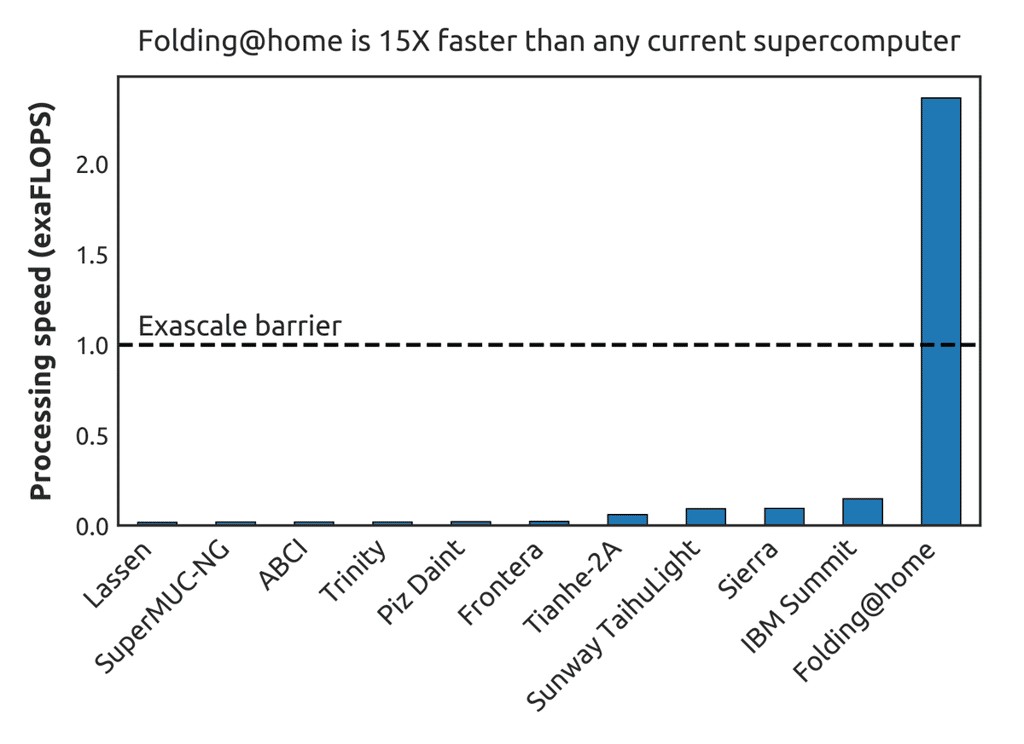
2,5 milliards de milliards d’opérations à virgule flottante par seconde
Et les contributeurs sont désormais nombreux car depuis ces dernières semaines la puissance de calcul de Folding@home a augmenté de façon vertigineuse au point de damer le pion aux grandes puissances qui faisaient la course pour dépasser le murmur de l'exaFLOPS, c'est-à-dire un seuil de vitessevitesse de calcul correspondant à l'exécution d'un milliard de milliards d'« opérations à virgule flottante par seconde » (floating point operations per second ou FLOPS, en anglais)
Les Chinois espéraient passer ce mur de l'exaFLOPS à l'horizon 2021 au centre National University of Defense Technology de Guangzhou et les Américains également avec l'AuroraAurora du DOE (Department of Energy), construit par Cray pour le compte du Laboratoire national d'Argonne (à Chicago). Rappelons que le mur du pétaFLOPS (un million de milliards de FLOPS) avait été, lui, franchi par le RoadRunner d'IBMIBM en 2008.
Mais aujourd'hui, Folding@home a déjà atteint les 2,5 exaFLOPS, soit une puissance supérieure à celle que l'on obtiendrait si on réunissait les 500 supercalculateurssupercalculateurs les plus puissants au monde. Et ce n'est sans doute pas fini ! La noosphère est en tout cas bel et bien entrée dans l'ère exaflopique...
Le Cern contribue de plusieurs façons à la lutte contre la pandémie. Pour obtenir une traduction en français assez fidèle, cliquez sur le rectangle blanc en bas à droite. Les sous-titres en anglais devraient alors apparaître. Cliquez ensuite sur l'écrou à droite du rectangle, puis sur « Sous-titres » et enfin sur « Traduire automatiquement ». Choisissez « Français ». © Cern
Ce qu’il faut
retenir
- Un traitement possible contre le Covid-19 suppose la modélisation de la structure et de la dynamique d’une protéine à la surface du Covid-19. Elle intervient dans les dégâts causés par la maladie en se liant à une protéine réceptrice sur une cellule pulmonaire. Un traitement consisterait donc à trouver un anticorps thérapeutique, c’est-à-dire un type de protéine qui peut empêcher la protéine virale de se lier à son récepteur, empêchant ainsi le virus d'infecter la cellule pulmonaire.
- L'opération demande une grande puissance de calcul qui est atteinte à l'échelle de la Planète bleue grâce au logiciel Folding@home qui permet de faire du calcul distribué, lequel consiste à découper en petites unités indépendantes des calculs à effectuer, pour les répartir ensuite sur des ordinateurs personnels (Mac ou PC).
- Le Cern vient de joindre à ces ordinateurs certaines des machines de son centre de calcul et aujourd’hui Folding@home a déjà atteint les 2,5 exaFLOPS, soit une puissance supérieure à celle que l'on obtiendrait si on réunissait les 500 supercalculateurs les plus puissants au monde.