Des chercheurs de l’école polytechnique de Zurich ont réécrit informatiquement le génome d’une bactérie afin qu’il soit plus simple à produire chimiquement en laboratoire. Cette innovation pourrait révolutionner les biotechnologies.
au sommaire
Un nouveau génome vient de faire son entrée dans la banque de données des génomes connus : celui de la bactérie Caulobacter ethensis-2.0, le premier génome entièrement créé par ordinateurordinateur. Mais si ce génome est bien réel, l'être vivant correspondant n'existe pas encore... Cette recherche s'inscrit dans la lignée des travaux des dernières décennies sur les génomes de synthèse.
Au début des années 2000, des chercheurs ont recréé en laboratoire des génomes de virus. Aux États-Unis, l'équipe de Craig Venter a travaillé sur de petites bactéries, Mycoplasma genitalium et M. mycoides. Ce laboratoire a créé une bactérie minimaliste à partir du génome de M. mycoides, avec uniquement 473 gènes « essentiels ».
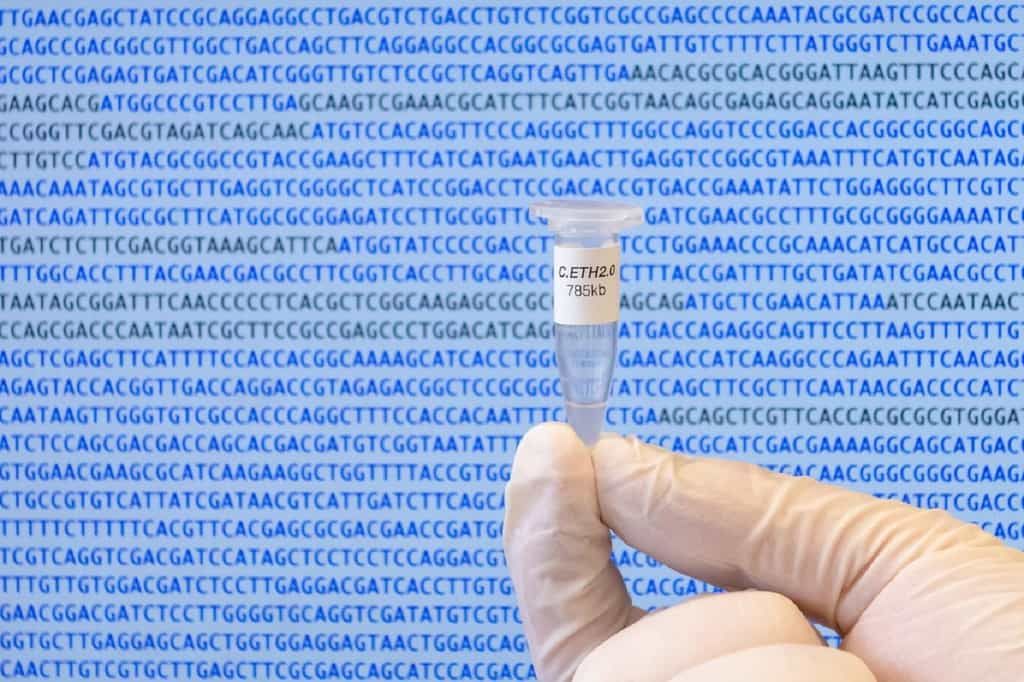
Ici, le génome de C. ethensis-2.0 a été inventé à partir de Caulobacter crescentus, une bactérie modèle inoffensive qui vit en eau douceeau douce, dans des rivières ou des lacs. Son génome contient 785.000 bases (785 kb) et 4.000 gènes, dont seulement 680 gènes sont indispensables à sa survie dans les conditions du laboratoire.
Les chercheurs de l'École polytechnique fédérale de Zurich sont donc partis du génome minimal de C. crescentus et ont voulu le synthétiser sous la forme d'un anneau d'ADN. Au départ, les chercheurs ont fabriqué 236 fragments de génome qu'ils ont voulu assembler. Mais, techniquement, les génomes « naturels » sont souvent difficiles à synthétiser, comme l'a expliqué le chimiste Mathias Christen dans un communiqué : « La synthèse de ces segments n'est pas toujours facile. Les moléculesmolécules d'ADN possèdent non seulement la capacité de coller à d'autres molécules d'ADN, mais, en fonction de la séquence, elles peuvent également se tordre en boucles et en nœudsnœuds, ce qui peut entraver le processus de production ou rendre la fabrication impossible. »
Des gènes réécrits et toujours fonctionnels
Les chercheurs ont voulu optimiser ce génome avec un algorithme informatique qui l'a réécrit pour simplifier les étapes de fabrication et d'assemblage du génome. Par exemple, les chercheurs ont supprimé des régions riches en GC car de telles séquences entravent la synthèse chimique. Ils ont enlevé un millier de sites où des enzymes de restrictionenzymes de restriction pouvaient couper l'ADN, ceci pour optimiser l'assemblage ultérieur du génome. Les chercheurs ont aussi supprimé des séquences qui ne codaient pas pour des protéinesprotéines.
En même temps, il ne fallait pas que le message protéique change. Aussi, les chercheurs se sont appuyés sur la redondance du code génétiquecode génétique : un même acide aminéacide aminé peut être codé de différentes manières. Grâce à l'algorithme, les chercheurs ont créé une séquence optimale, en remplaçant environ 133.000 nucléotidesnucléotides, changeant plus de 123.000 codonscodons.
Le saviez-vous ?
Le génome d’une cellule, sous forme d’ADN, est constitué d’une succession de nucléotides, les « lettres » du message. L’ADN est transcrit en ARN messager (ARNm) qui sert à la fabrication des protéines. Un triplet de nucléotides, ou codon, code pour un acide aminé de la chaîne protéique. La correspondance se fait grâce au code génétique qui est redondant : un même acide aminé peut être codé par plusieurs codons.
Cependant, si l'on change un nucléotide, on peut quand même modifier la structure de l'ARNmARNm. Ces gènes réécrits étaient-ils fonctionnels ? Pour le savoir, les chercheurs ont créé des bactéries contenant à la fois le génome naturel de Caulobacter et des fragments du génome synthétique, afin de tester les gènes artificiels. Résultat : plus de 80 % des gènes de C. ethensis-2.0 étaient fonctionnels, et 98 gènes avaient perdu leur fonction.
Cette découverte parue dans PNAS est une innovation intéressante pour le secteur des biotechnologies. Pour Beat Christen, professeur de biologie et frère de Mathias, « il sera bientôt possible de produire des cellules bactériennes fonctionnelles avec un tel génome. » Ces micro-organismesmicro-organismes synthétiques pourraient servir à produire des molécules comme des médicaments, des vitaminesvitamines. Mais la création de tels organismes vivants soulève aussi des questions éthiques : « Aussi prometteurs que puissent être les résultats de la recherche et les applicationsapplications possibles, ils exigent une discussion en profondeur de la société sur les objectifs pour lesquels cette technologie peut être utilisée et, en même temps, sur les moyens de prévenir les abus », prévient-il.
Ce qu’il faut
retenir
- Des chercheurs suisses ont réécrit le génome d’une bactérie grâce à un algorithme.
- L’objectif était de simplifier la synthèse chimique du génome.
- La plupart des gènes réécrits étaient fonctionnels.