au sommaire
L'information génétique contenue dans l'ADN est déjà une forme de mémoire biologique. Mais les chercheurs veulent maintenant créer des logiciels à intégrer dans le monde vivant. La route est encore longue. Pour preuve, dans cette expérience pionnière, il fallait tout de même une heure avant que les recombinases aient changé l'orientation de l'ADN. Une éternité à côté des nanosecondes suffisantes pour une telle tâche dans un ordinateur. © Maurizio de Angelis, Wellcome Images, Flickr, cc by nc nd 2.0
La performance pourrait paraître sommaire à côté de celles réalisées par nos ordinateursordinateurs, mais il faut bien un début à tout. La biologie synthétique n'en est qu'à ses prémices... On vient de franchir une nouvelle étape dans la programmation du vivant en développant une forme de mémoire réinscriptible d'un bit encodée dans un brin d'ADN, comme expliqué dans les Pnas. À terme, cette prouesse originale pourrait se traduire par de nombreuses applicationsapplications biomédicales et servir par exemple de traitement contre le cancer, ou à cerner les mécanismes de vieillissement cellulaire.
Cette notion de logiciellogiciel intégré dans le vivant n'est pas nouvelle. De précédents travaux avaient permis d'insérer des dispositifs d'ADN que l'on pouvait activer ou désactiver à l'intérieur de la cellule. D'autres étaient parvenus à insérer un compteur permettant de dénombrer les occurrences d'un événement particulier, comme la division cellulaire. Mais une fois le dispositif mis en place, il n'était plus possible de lui donner de contrordre, c'est-à-dire d'effacer la mémoire et de la remplacer par une nouvelle, comme c'est le cas dans nos machines.
De l’ADN-R à l’ADN-RW
Cette performance vient d'être réalisée par des chercheurs de l'université Stanford, en Californie, qui sont parvenus à activer leur nanodispositif à plusieurs reprises, jusqu'à 16 fois. Mais en quoi consiste-t-il ?

Les bactériophages sont des virus qui infectent les bactéries et intègrent leur ADN dans celui de leur hôte. Ils disposent donc des enzymes nécessaires pour insérer leur génome dans le chromosome bactérien. Les chercheurs ont profité de ces propriétés. © Anna Tanczos, Wellcome Images, Flickr, cc by nc nd 2.0
Dans cette expérience, un brin d'ADN possède à chacune de ses extrémités des séquences nucléotidiques particulières, reconnues par des enzymesenzymes appelées recombinases, issues d'un bactériophage, un virus qui comme son nom l'indique infecte des bactéries. Ces recombinases clivent l'ADN, le retourne et le replace à l'envers sur le chromosome. Grâce à d'autres enzymes, des sérines intégrases ou excisionases, l'activité des recombinases a été contrôlée, de manière à les forcer à orienter le brin d'ADN dans un sens puis dans l'autre.
Combien de temps pour augmenter la mémoire à ADN ?
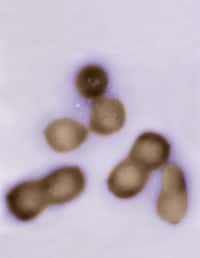
Pour vérifier l'efficacité de leur dispositif, les auteurs l'ont testé dans la bactériebactérie Escherichia coliEscherichia coli, célèbre car très utilisée en biologie mais aussi parce qu'elle a causé une épidémie mortelle l'année passée en Europe et surtout en Allemagne. L'ADN était ainsi conçu qu'il devenait fluorescent en rouge ou en vert en fonction du sens dans lequel il était orienté. Les bactéries changeaient effectivement de couleurcouleur.
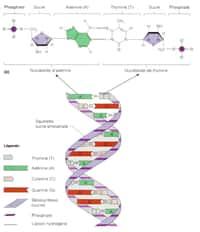
Ce système se rapproche donc du langage de nos ordinateurs, les deux orientations possibles pouvant correspondre aux 0 et 1 du binairebinaire. Cette opération consiste donc en l'intégration dans la cellule d'une mémoire de 1 bit. Les chercheurs espèrent désormais passer à l'octetoctet (soit 8 bits), ce qui peut demander des années encore.
En effet, la biologie synthétique n'est pas au point. Il leur a fallu 750 versions pour enfin obtenir ce qu'ils souhaitaient. En langage informatique, on dirait qu'ils ont dû réparer 750 bugsbugs. Et comme les séquences ADN ne s'écrivent pas aussi facilement que les lignes de code utilisées dans les logiciels, ce travail leur a demandé trois ans. Combien de temps sera-t-il nécessaire pour développer un dispositif huit fois plus imposant ?