au sommaire
Évolution du virus A (H5N1)
Les premiers cas avérés d'infection humaine grave voire mortelle due aux virus HPAI datent de 1997, à Hong Kong, pendant les épizootiesépizooties de grippe aviairegrippe aviaire qui dévastent les élevages de volaille. L'élimination de toutes les volailles met fin à la flambée. Bien que le virus A (H5N1H5N1) continue de circuler chez les oiseaux d'élevage, aucun nouveau cas humain ne sera signalé entre 1999 et février 2003.

Virus A (H5N1). © acces-inrp.fr
Le séquençageséquençage des segments viraux effectué sur les isolats cliniques A (H5N1) de 1997 montre la proximité avec les virus qui circulent chez les volailles et les oiseaux sauvages et met en évidence des facteurs de virulence que l'on retrouvera ultérieurement au cours des flambées de 2003 à 2005 (Beigel, Farrar et al. 2005 11) :
- les acides aminésacides aminés basiques multiples au niveau du site de clivageclivage de l'hémagglutininehémagglutinine lui permettent d'être activée par de nombreuses protéasesprotéases cellulaires ;
- la substitution de la glutamineglutamine en position 627 par la lysinelysine, au niveau de la protéineprotéine PB2 augmente la capacité réplicative du virus ;
- la substitution de l'asparagineasparagine en position 92 par la glutamine, au niveau de la protéine NS1, accroît la résistancerésistance du virus à l'activité antivirale des interféronsinterférons et du facteur nécrose tumorale α in vitroin vitro.
Les études antigéniques et phylogénétiquesphylogénétiques d'isolats A (H5N1) aviaires et humains prélevés en 2004 et 2005 en Asie (WHO Global InfluenzaInfluenza Program 2005 98), montrent une proximité étroite du gènegène de l'hémagglutinine des spécimens humains avec l'HA des virus aviaires (taux de divergence <=1%) et l'existence de deux linéages (ou cladesclades) différents pour l'HA, appelés clade 1 et clade 2 :
- le clade 1 est isolé chez les oiseaux et l'homme au Vietnam, en Thaïlande et au Cambodge, uniquement chez les oiseaux au Laos et en Malaisie ;
- le clade 2 est isolé chez les oiseaux uniquement en Chine, en Indonésie, au Japon et en Corée du sud ;
- ils sont différents des virus isolés chez les oiseaux et l'homme à Hong Kong en 2003 et 1997 qui appartiennent à des clades différents : 1' et 3 respectivement.
La comparaison des séquences d'acides aminés de l'HA des isolats humains de clade 1 et 2 avec ceux des cas mortels de 1997 et 2003 à Hong Kong, montre des variations au niveau de l'HA des virus 2004-2005 (WHO Global Influenza Program 2005 98) :
- la substitution de la sérinesérine vers la leucineleucine à la position 126 est associée à une modification de la capacité de fixation du virus au récepteur cellulaire ;
- la substitution de l'adénosineadénosine vers la tyrosinetyrosine à la position 156, entraîne une glycosylation de l'asparagine à la position 154. Cette modification est habituellement associée à une adaptation du virus aux oiseaux terrestres et une accentuation de la virulence chez ces oiseaux.
De nombreuses modifications des acides aminés de l'hémagglutinine apparaissent dans les trois premiers mois de 2005 par rapport aux isolats de 2004. Les plus fréquentes sont localisées près du site de fixation au récepteur cellulaire (WHO Global Influenza Program 2005 98).
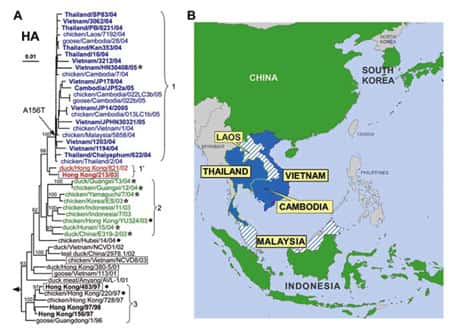
Relations phylogénétiques entre les gènes de l'hémagglutinine des virus influenza H5N1 (les clades 1, 1´, 2 et 3 respectivement en bleu, rouge, vert et noir) et à droite, leur répartition géographique en Asie. © Ruben O. Donis, Influenza Branch, DVRD, NCID, Centers for Disease Control and Prevention; Emerging Infectious Diseas, 2005 Oct
Arbre phylogénétique des gènes de la neuraminidase NA
L'arbre phylogénétiquearbre phylogénétique des gènes de la neuraminidaseneuraminidase NA est semblable à celui des gènes de l'HA, avec une évolution parallèle des deux gènes de l'enveloppe virale. Les gènes des isolats de Thaïlande semblent diverger des gènes de virus isolés au Vietnam, pour former un groupe distinct. Les gènes de la NA des isolats humains et aviaires de 2003 à 2005 ainsi que ceux du clade 3 présentent des délétionsdélétions au niveau de la tige de l'enzyme, correspondant aux acides aminés de la position 49 à 68 dans les clades 1 et 2, de la positions 54 à 72 dans le clade 3. Ces délétions favoriseraient la rétention des virionsvirions au niveau de la membrane plasmiquemembrane plasmique pour contrebalancer la faiblesse de la liaison entre l'HA et l'acide sialique induite par la glycosylation de l'acide aminé en position 154 récemment acquise (WHO Global Influenza Program 2005 98).
Arbre phylogénétique des gènes de la protéine M2
L'arbre phylogénétique des gènes de la protéine M2 évolue de façon parallèle à celui des gènes de la HA. La séquence d'acides aminés de M2 des virus du clade 1 et du clade 1' (A/Hong Kong/213/03) révèle une substitution de la sérine vers l'asparagine au résidu 31 ; cette substitution confère la résistance aux adamantanes. Les virus de clade 1 de 2004 et 2005, cultivés en présence de rimantadine se répliquent aussi efficacement qu'en l'absence de l'antiviralantiviral dans le milieu de culture (WHO Global Influenza Program 2005 98).
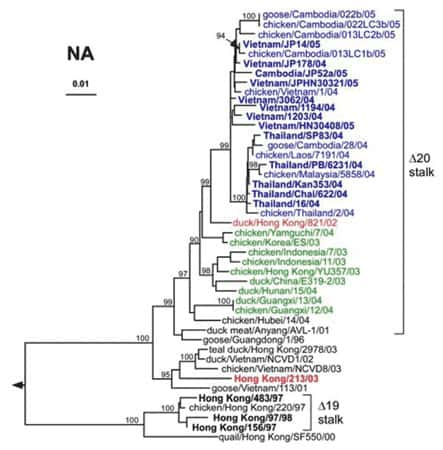
Relations phylogénétiques entre les gènes de la neuraminidase N1 des virus influenza H5N1 (les clades 1, 1´, et 2 respectivement en bleu, rouge et vert). © Ruben O. Donis, Influenza Branch, DVRD, NCID, Centers for Disease Control and Prevention; Emerging Infectious Diseas, 2005 Oct
Tous les gènes sont d'origine aviaire
La caractérisation complète des gènes des virus humains circulant en Asie de 2003 à 2005 confirme l'origine aviaire de tous les gènes et l'absence de tout réassortiment avec des gènes d'origine humaine.
Depuis 1997, la circulation du virus A (H5N1) dans de nombreuses espècesespèces aviaires, s'est accompagnée de nombreux réassortiments et a favorisé l'apparition de nombreux génotypesgénotypes. La phylogénie a permis de tracer l'origine du virus A (H5N1) hautement pathogènepathogène asiatique, en comparant des isolats aviaires et humains d'Indonésie, de Thaïlande, du Vietnam, avec des prélèvements effectués au cours de la surveillance systématique sur les marchés de Hong Kong et en Chine, de 2000 à 2004. Les gènes codant pour la HA et la NA dérivent de la souche Goose/Guangdong/1/96, isolée en Chine en 1996. Les six gènes codant pour les protéines internes sont issus de multiples réassortiments (Sims, Domenech et al. 2005 87).
Depuis janvier 2002, le génotype Z est le type dominant en Chine du sud : caractérisé par la présence de délétions au niveau de la tige de la neuraminidase, et de délétions de cinq acides aminés (position 80-84) au niveau de la protéine NS1. Tous les virus responsables des flambées en Indonésie, Thaïlande et au Vietnam, fin 2003 et 2004 sont de génotype Z (Li, Guan et al. 2004 54) (Puthavathana, Auewarakul et al. 2005 78). La caractérisation du virus A (H5N1) isolé chez l'homme en 2003, à Hong Kong montre l'absence de délétion au niveau de la tige de la neuraminidase et l'absence de site de glycosylation supplémentaire à la tête de l'hémagglutinine ; ces caractéristiques sont typiquement associées à l'adaptation du virus aux oiseaux terrestres (Shinya, Hatta et al. 2005 85).