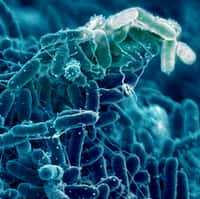
Des chercheurs viennent d’établir la première base génétique des milliers de virus présents dans notre intestin. Une étude riche d’enseignements, qui montre notamment que notre « virome » évolue fortement selon l’âge ou le mode d’alimentation et constitue une signature propre à chaque individu.
au sommaire
Chacun connaît désormais le rôle du microbiote humain. Ces millions de bactéries peuplant notre intestin jouent un rôle prépondérant dans notre santé : immunité, digestion, obésité, cancer, maladies cardiovasculairesmaladies cardiovasculaires, diabètediabète, hypertensionhypertension, métabolisme, dépression... La composition de notre microbiote influerait même sur les performances sportives et la personnalité. Mais les bactéries ne sont pas les seules à séjourner dans notre intestin : elles cohabitent avec des parasitesparasites et des virusvirus. Autant les bactéries intestinales sont étudiées sous toutes les coutures, autant le monde des virus reste largement inconnu. Et pour cause : « Contrairement aux bactéries, les virus ne partagent pas de séquence génétiquegénétique commune », explique OlivierOlivier Zablocki, microbiologiste à l'université d'État de l'Ohio et auteur avec ses collègues d'une nouvelle étude parue dans Cell Host & Microbe. « Une vaste partie du viromevirome [nom donné à l'ensemble des virus présents dans l'intestin humain] reste donc inexplorée, que l'on appelle souvent "matière noirematière noire" ».
Une base de données de 33.000 virus
Durant plus de 10 ans, les chercheurs de l'université d'État de l'Ohio ont patiemment assemblé les données des virus déjà connus issues de différentes études, puis utilisé un logiciellogiciel d'intelligence artificielleintelligence artificielle pour détecter et identifier les génomesgénomes de virus inconnus. Bilan : une énorme base de donnéesbase de données contenant pas moins de 33.242 virus des intestins récoltés sur près de 2.000 individus dans 16 pays, que les chercheurs ont nommé Gut Virome Database (GVD).
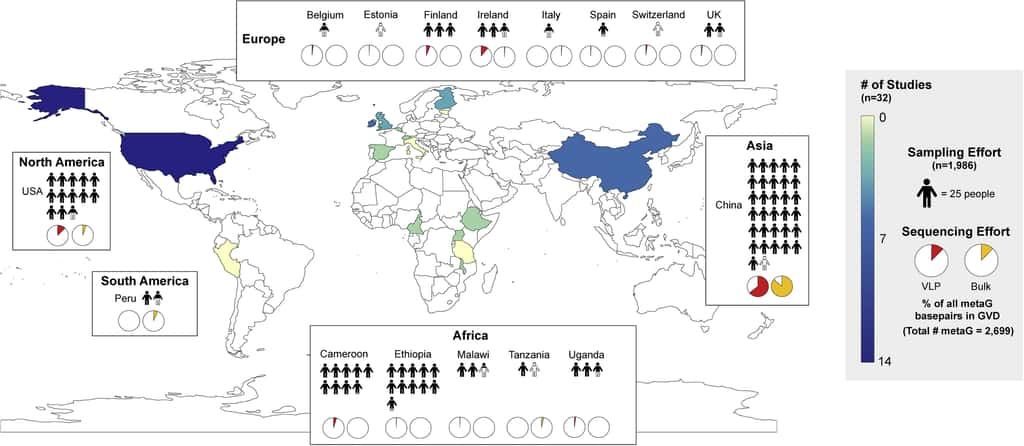
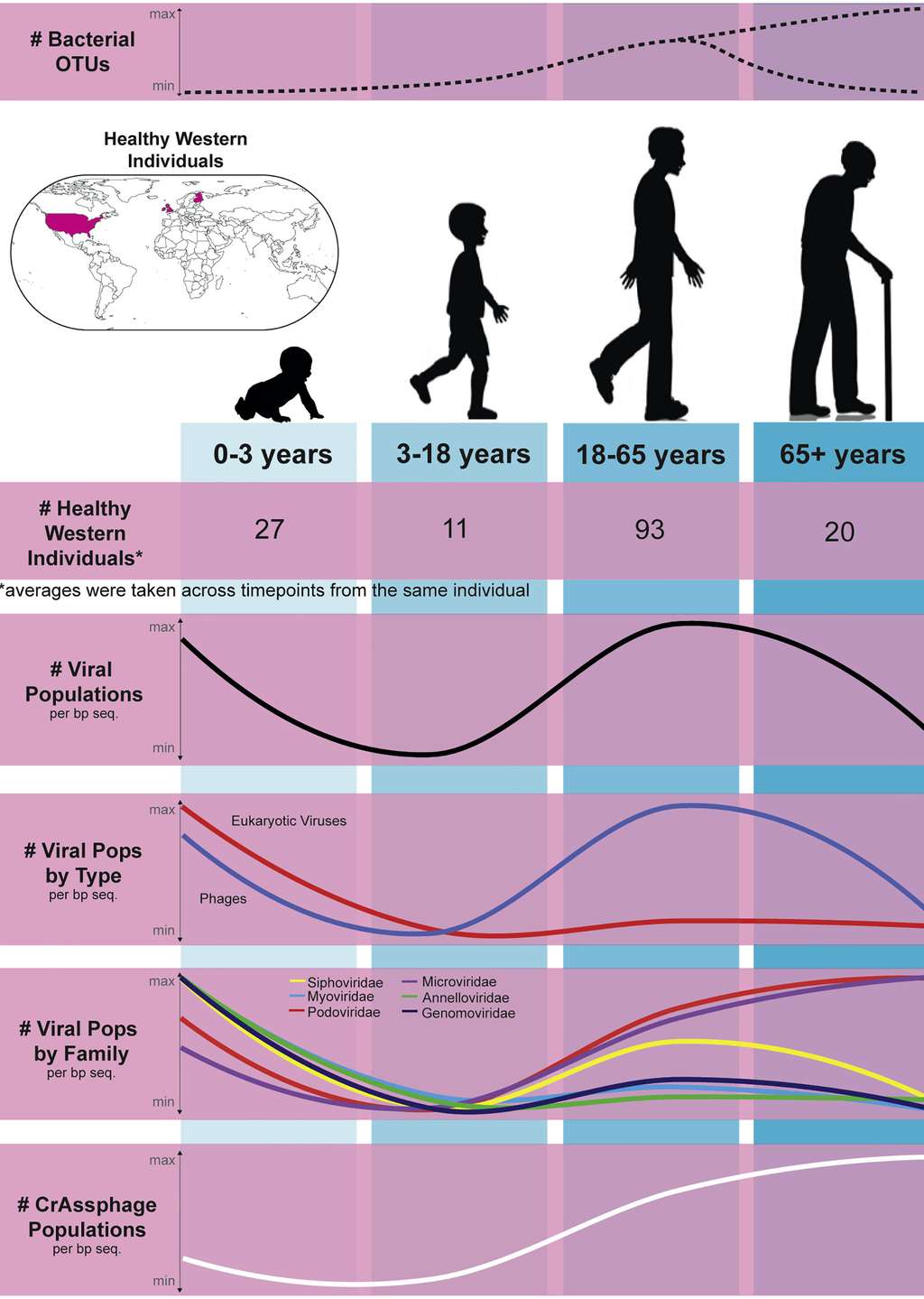
Bien que quelques populations virales soient partagées au sein d'un sous-ensemble de personnes, il n'existe pas de groupe de virus intestinaux communs à tous les humains. En fait, chaque individu possède sa propre « signature virale » du microbiote, un peu comme une empreinte digitaleempreinte digitale. Les chercheurs ont malgré tout pu tirer quelques tendances générales.
- L'âge est un facteur prépondérant dans la diversité de virus : cette dernière augmente considérablement de l'enfance jusqu'à l'âge adulte puis diminue après 65 ans. Logique, car cela suit l'évolution de la diversité bactérienne intestinale. À une exception près : les intestins des nourrissons qui regorgent de divers types de virus, mais contiennent peu de variétés de bactéries. Ce serait dû à leur système immunitairesystème immunitaire encore sous-développé, moins à même de répondre aux attaques des virus.
- Les personnes vivant dans les pays occidentaux ont une moins grande diversité de virus dans leur intestin. Une tendance que l'on observe là encore aussi pour le microbiote bactérien, et qui confirme que le mode d’alimentation et l'environnement des Occidentaux appauvrissent la diversité du microbiote. Or, « une règle générale en matière d'écologieécologie est qu'une plus grande diversité conduit à un écosystèmeécosystème plus sain, indique Ann Gregory, coauteur de l'étude. Les individus en bonne santé ont tendance à avoir une plus grande diversité de virus, ce qui indique que ceux-ci ont potentiellement un rôle bénéfique ».
Virome : 98 % de phages dans l’intestin
Mais d'ailleurs que font tous ces virus dans notre intestin ? Rappelons qu'un virus ne subsiste pas par lui-même mais doit obligatoirement infecter un hôte pour survivre. De fait, presque tous les virus détectés (97,7 %) sont des phages, c'est-à-dire des virus qui infectent les bactéries. La plupart tuent leur hôte mais les chercheurs ont aussi découvert des phagesphages qui coexistent avec les bactéries hôtes et produisent même des gènesgènes qui les aident à rivaliser et à survivre. Une sorte de symbiose gagnant-gagnant.
À présent, la base de données va pouvoir servir de plusieurs façons. Les chercheurs envisagent, par exemple, d'utiliser ces phages pour détruire les bactéries nuisibles de l'intestin ou ailleurs, en particulier celles résistantes aux antibiotiquesantibiotiques classiques. L'étude du virome pourrait aussi servir à expliquer l'origine de certaines maladies ou la susceptibilité des gens à telle ou telle pathologiepathologie, par exemple les symptômes gastro-intestinaux ressentis par certains des patients atteints de Covid-19Covid-19. Pour des plus amples investigations, les chercheurs en appellent à élargir encore la base de données. Du travail en perspective.
Microbiote : près de 2.000 nouvelles espèces de bactéries découvertes
Article de Marie-Céline RayMarie-Céline Ray publié le 14/02/2019
Des chercheurs britanniques ont reconstruit le génome de bactéries du microbiote grâce à des méthodes informatiques. Ils ont ainsi découvert dans l'intestin humain près de 2.000 nouvelles espècesespèces de bactéries qui n'avaient pas encore été cultivées en laboratoire.
Notre intestin est peuplé de milliers de micro-organismes qui forment le microbiote et jouent un rôle important dans la digestion et l'immunitéimmunité. Certaines pathologies, comme des maladies inflammatoires chroniques de l'intestin, sont associées à un déséquilibre de la flore intestinaleflore intestinale. Mais beaucoup de ces bactéries sont encore méconnues car il est compliqué de les cultiver au laboratoire. D'où l'idée d'utiliser la bio-informatique pour identifier de nouvelles espèces dans le microbiote intestinal.
C'est ce qu'explique, dans un communiqué, Robert Finn, l'un des auteurs de cette recherche : « Les méthodes informatiques nous permettent de comprendre des bactéries que nous ne pouvons pas encore cultiver en laboratoire. Les chercheurs sont maintenant à un stade où ils peuvent utiliser une gamme d'outils informatiques pour compléter et parfois guider les travaux de laboratoire, afin de découvrir de nouvelles perspectives sur l'intestin humain. »
Cette étude, parue dans la revue Nature, a été effectuée par des chercheurs de l'Institut européen de bio-informatique (EMBL-EBI) et du Wellcome Sanger Institute. À partir de 11.850 microbiomesmicrobiomes d'intestins humains, les chercheurs ont reconstruit plus de 92.000 génomes, grâce à la métagénomiquemétagénomique.
Des micro-organismes difficiles à cultiver au laboratoire
Les chercheurs ont ainsi identifié 1.952 nouvelles espèces de bactéries, qui n'avaient pas été cultivées. Environ la moitié de ces espèces ne pouvaient pas être classées dans des genres déjà connus. Ces nouvelles espèces augmentent d'une manière considérable le nombre d'espèces connues dans l'intestin humain, même si ces espèces étaient peu fréquentes dans les intestins bien étudiés.

Les auteurs notent également que cette analyse métagénomique du microbiote a aussi mis en évidence des génomes appartenant à des organismes non-bactériens et qui mériteraient d'être étudiés : virus, archéesarchées et eucaryoteseucaryotes.
L'étude s'est aussi intéressée aux différences régionales dans la flore intestinale. « Nous constatons que de nombreuses espèces bactériennes identiques apparaissent dans les données provenant des populations européennes et nord-américaines. » D'après le chercheur, les quelques données disponibles sur d'autres populations d'Amérique du Sud ou d'Afrique, ont révélé une diversité absente des autres populations. Mais les informations sur la flore intestinale d'habitants d'Amérique du Sud, d'Asie ou d'Afrique sont encore insuffisantes.
Ces résultats améliorent nos connaissances sur les micro-organismesmicro-organismes qui peuplent les intestins des habitants d'Amérique du Nord et d'Europe. De telles données pourraient, à l'avenir, aider à mieux diagnostiquer et traiter des maladies liées au microbiote intestinal.