au sommaire
Streptococcus thermophilus © Inra
Le yaourtyaourt est obtenu par la fermentationfermentation du lait, à l'aide de deux bactériesbactéries, un lactobacille, dénommé bulgaricus et un streptocoquestreptocoque, dit thermophilus, qui agissent ensemble. Les deux bactéries se retrouvent vivantes dans le yaourt et donc, dans la bouche du gourmet qui le consomme. Et comme les amateurs de yaourt sont nombreux de par le monde, la quantité de bactéries que nous ingurgitons est véritablement astronomique - on estime que l'humanité consomme tous les ans un milliard de milliard de milliards (10 puissance 21) de cellules vivantes de Streptococcus thermophilus.
L'analyse a démontré très clairement que S. thermophilus a perdu l'essentiel des gènesgènes reconnus importants dans le pouvoir pathogènepathogène de S. pneumoniae ou S. pyogenes. Ces gènes, liés à la virulence ("virulence related genes") des deux bactéries, sont soit présents, mais dans une forme non-fonctionnelle à cause de mutations qui les inactivent, soit totalement absents chez S. thermophilus.
Les chercheurs ont confirmé ces mêmes mutations ou absences de gènes dans la séquence du génomegénome de deux souches différentes de S. thermophilus isolées du yaourt, qui ont divergé et évolué séparément depuis au moins 3 000 ans.
L'inactivation ou la perte de ces gènes se sont donc produites lors de l'adaptation de S. thermophilus à un environnement nouveau, le lait, où ils n'étaient plus d'aucune utilité (le début de la fermentation du lait par l'homme est estimé à il y a 7 000 ans). D'une façon semblable, bien des gènes nécessaires à l'utilisation des sucressucres absents du lait sont fonctionnels chez les streptocoques pathogènes mais ne sont plus actifs chez S. thermophilus. En tout, quelque 10% des gènes de cette bactérie ne sont pas fonctionnels et pourraient donc bien être en passe d'être entièrement éliminés.
La perte et l'inactivation des gènes liés à la virulence rendent donc totalement invraisemblable la possibilité que S. thermophilus mette en danger la santé de l'homme - les amateurs du yaourt peuvent continuer de s'en délecter.
L'analyse du génome a montré d'autre part, que l'adaptation au lait a entraîné l'acquisition de gènes utiles à S. thermophilus dans sa nouvelle niche écologique. Ainsi, cette bactérie peut tirer l'énergie nécessaire pour sa croissance à partir du sucre du lait, le lactoselactose, ce que les cousins pathogènes de la bactérie du yaourt ne peuvent pas faire.
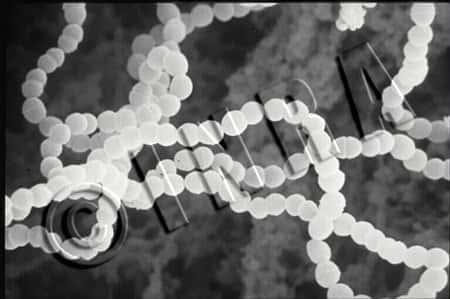
Et comment S. thermophilus a-t-il pu se procurer des gènes utiles dans le lait ? En partie, grâce à des bactéries avec lesquelles il partage la niche écologique. En effet, dans le génome de S. thermophilus se trouvent les gènes nécessaires à la synthèse de la méthionineméthionine, un acide aminéacide aminé rare dans le lait, dont plusieurs proviennent de l'autre bactérie du yaourt, Lactobacillus bulgaricus ! La microscopie révèle que les deux bactéries vivent en étroite association, les coques rondes étant accolées aux bacillesbacilles longilignes, à l'image des perles le long du fil d'un collier, ce qui favorise certainement les échanges de gènes entre les deux.
La proximité écologique serait donc bien plus importante que la proximité phylogénétiquephylogénétique pour le flux génétiquegénétique entre les espècesespèces, étant donné que les lactobacilles et les streptocoques semblent diverger depuis 1,5 à 2 milliards d'années, une période comparable à celle des organismes multicellulaires, précurseurs des plantes et des animaux, apparus il y 1,2 milliards d'années.