au sommaire
Si le siècle précédent a été marqué par la révolution de la physique et les technologies qu'elle a engendrées, le XXIe siècle devrait être celui de la biologie, des neurosciences et de la nanotechnologienanotechnologie. Ces révolutions annoncées ne seraient rien sans l'aide des ordinateursordinateurs et les possibilités qu'ils ouvrent dans le domaine que l'on appelle aujourd'hui le Big data. Une médecine spécialisée tenant compte des spécificités génétiquesgénétiques de chaque individu est particulièrement attendue.
Cependant, encore faut-il pour cela disposer du génomegénome des patients et pouvoir l'interpréter. Au final, cela se réduit à deux conditions : pouvoir réaliser le séquençage de l’ADN à bas prix et, très rapidement, pouvoir utiliser un grand nombre de génomes pour en tirer des conclusions quant aux particularités des personnes et les implications pour les maintenir en bonne santé. Cette dernière condition implique qu'il soit possible de faire des recherches avec les techniques du Big dataBig data, ce qui suppose d'ailleurs la création d'un Internet de l'ADN.
Il est question, depuis plus d'une décennie, du matériau miracle qu'est le graphènegraphène. Ses propriétés exceptionnelles tant du point de vue de l'électronique que de la mécanique ont valu le prix Nobel de physique à ses découvreurs et l'attribution d'un budget d'un milliard d’euros pour les recherches le concernant dans le cadre de l'Union européenne.
D'autres laboratoires sur la planète ne sont pas en reste comme le montre une publication déposée sur arXiv par des membres du National Institute of Standards and Technology (NISTNIST), aux États-Unis. Elle concerne la possibilité d'utiliser le graphène pour faire encore baisser les coûts du séquençageséquençage de l'ADNADN tout en l'effectuant en un temps raisonnable.
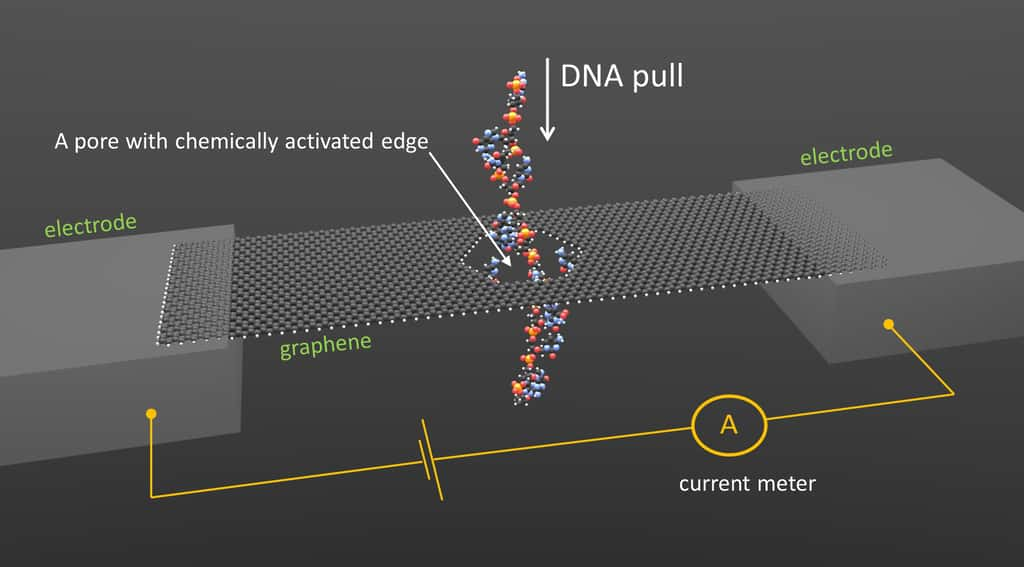
Un nanoruban de graphène est collé entre deux électrodes et soumis à une différence de potentiel alors qu'un ampèremètre mesure des variations de courants. Ces variations se produisent lorsqu'un brin d'ADN (DNA, en anglais sur l'image) traverse un pore portant des bases bien spécifiques de l'ADN (A pore with chemically activated edge). © Smolyanitsky/NIST
Des nanorubans de graphène pour détecter les bases de l'ADN
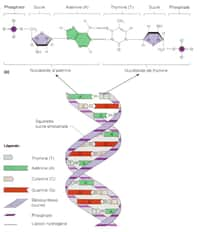
Ces chercheurs états-uniens ne sont pas les premiers à considérer cette possibilité. Surtout, elle n'est encore que théorique car reposant essentiellement sur des simulations numériquessimulations numériques. Pourtant, si les chercheurs ont raison, il devrait être possible d'identifier 66 millions de bases de l'ADN par seconde, désignées par les célèbres lettres A, C, G, T et U (l'adénineadénine, la cytosinecytosine, la guanineguanine, la thyminethymine et l'uracileuracile), avec un taux de succès de 90 %.
L'idée de base pour réussir cet exploit est la suivante : tout commence par la fabrication des nanorubans découpés dans un feuillet de graphène dont les dimensions sont de 4,5 par 15,5 nanomètres, avec des nanopores dont le diamètre est de 2,5 nanomètres. Des bases d'un type donné sont attachées à un nanoruban en bordure des pores. Lorsqu'un brin d'ADN traverse ces pores, des liaisons hydrogène temporaires se forment entre une base du brin et celle qui lui correspond selon les règles habituelles dans ce brin mais, cette fois-ci, avec la base que porteporte un nanoruban. Il peut donc se former des paires cytosine-thymine et guanine-adénine par exemple. Le nanoruban est donc soumis à des contraintes au fur et à mesure que le brin d'ADN traverse le pore puis retourne à l'équilibre après que les liaisons hydrogène ont été brisées.
Ces variations de contraintes modifient le courant électriquecourant électrique produit en imposant une différence de potentiel aux bords des nanorubans (voir le dessin ci-dessus). On peut donc détecter le passage d'une base donnée dans un brin d'ADN à travers le pore. En empilant plusieurs nanorubans spécifiques à chaque base, il devient donc possible de séquencer l'ADN en mesurant les variations de courant traversant ces feuillets de graphène.
Selon les simulations sur ordinateur des chercheurs, tout ce processus peut se dérouler à température ambiante dans de l'eau. Quatre mesures indépendantes du même brin d'ADN permettent, en théorie, de reconstruire une séquence avec 99,99 % de succès. Peut-être n'est-il pas déraisonnable de penser que dans un avenir proche, avec cette technique, tout le monde pourra voir son ADN séquencé en quelques minutes n'importe où sur la planète avec un appareil de la taille d'un ordinateur portable. Quelles leçons tirerons-nous alors de la lecture du génome des individus ? Quels impacts cela aura-t-il sur la société ? Espérons que le rêve ne se transforme pas en cauchemar, ouvrant la porte du monde de Bienvenue à Gattaca.