au sommaire
Les mécanismes d'expression des gènes
Deux grandes étapes sont nécessaires afin d'obtenir une protéineprotéine à partir d'un gènegène. La transcriptiontranscription permet d'obtenir une copie du gène présent sur le chromosomechromosome, sous forme d'un ARN messagerARN messager (ARNm). La traduction est l'étape durant laquelle cet ARNm est lu par le ribosomeribosome, pour obtenir une protéine. Afin d'obtenir une protéine active chacune de ces deux étapes doit être contrôlée rigoureusement pour être effectuée fidèlement.
- 1.1 La traduction
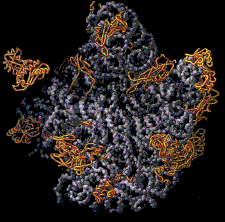
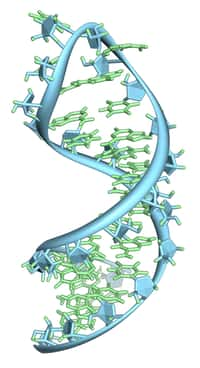
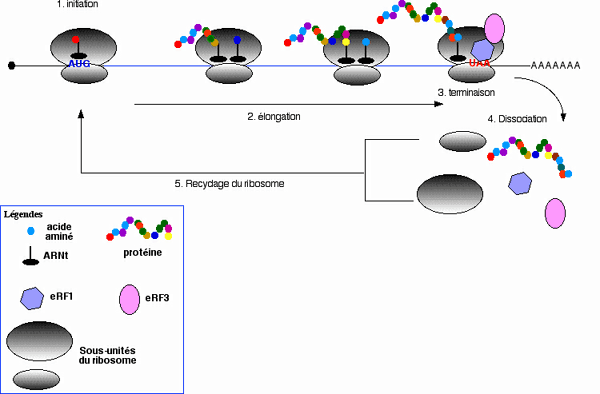
Longtemps négligée dans l'étude de la régulation des gènes, au profit de la transcription, la traduction a fait l'objet ces dernières années d'études intensives. La traduction correspond à la lecture de l'ARNm par le ribosome. Cette lecture s'effectue trois nucléotidesnucléotides par trois nucléotides (3 nucléotides = 1 codoncodon). Elle commence au codon initiateur (AUG), pour se terminer à l'un des trois codons stop (UAA, UAG ou UGA) (Figure 1). Au niveau de chaque codon, le ribosome incorpore un ARN de transfertARN de transfert (ARNt) chargé à son extrémité avec un acide aminéacide aminé. Cet ARNt possède une séquence complémentaire (anticodon) au codon présent dans le site de décodage du ribosome. L'appariement ARNt-ARNm permet de distinguer l'ARNt qui doit effectivement être incorporé d'un ARNt entré dans le ribosome par erreur (Figure 2). Une fois l'identité de l'ARNt vérifiée, la chaîne protéique en élongation est transférée sur le nouvel acide aminé, grâce à l'activité peptidyl-transferase du ribosome. Une fois le transfert effectué, le ribosome se déplace d'un codon (translocationtranslocation) et un nouveau cycle s'effectue jusqu'à ce que le ribosome rencontre un codon stop (figure 1). Lorsque le codon stop pénètre dans le ribosome, les facteurs de terminaison (eRF) vont provoquer l'arrêt de la synthèse protéique, le relargage de la protéine nouvellement synthétisée, et la dissociation des deux sous-unitéssous-unités du ribosome (figure 1).

Schéma général représentant les 5 grandes étapes de la traduction
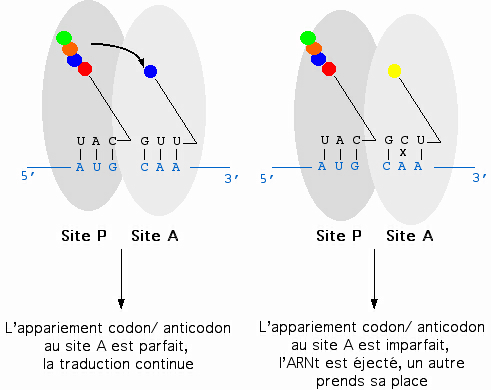
Représentation schématique des sites A et P du ribosome. L'ARNm est repésenté en bleu, les ARNt en noir, les acides aminés par des sphères colorées. L'ARNt pénètre dans le ribosome au site A. Si l'appariement codon/anticodon est correct, alors il y a transfert de la protéine en élongation sur le nouvel acide aminé apporté par l'ARNt. Dans le cas contraire, l'ARNt est éjecté du ribosome et un autre prend sa place.
La traduction est un point de régulation important de l'expression des gènes (1). Il a été montré que ces régulations pouvaient se dérouler au niveau de l'initiation de la traduction, de l'élongation (2, 3), mais aussi au niveau de la terminaison et ceci aussi bien chez les eucaryoteseucaryotes que les procaryotesprocaryotes (4, 5).
- 1.2 La terminaison de la traduction.
Les mécanismes moléculaires de cette dernière étape de l'expression des gènes ont récemment été élucidés chez les bactériesbactéries (6). En revanche chez les eucaryotes ces mécanismes demeurent encore mal compris. Dans les conditions normalesconditions normales l'efficacité de terminaison est de l'ordre de 99,7 %. Cependant il a clairement été démontré que cette efficacité était fortement dépendante de plusieurs paramètres :
A. La nature du codon stop. Les trois codons stop ne permettent pas la même efficacité de terminaison. Il est généralement admis que le codon UAA est le plus efficace, les codons UGA et UAG présentant une efficacité de terminaison légèrement plus faible. Il n'existe pas naturellement dans la cellule des ARNt possédant un anticodon parfaitement adapté pour reconnaître les codons stop. Cependant, l'hypothèse la plus probable pour expliquer ces différences d'efficacité entre les différents codons stop est l'existence dans la cellule d'un nombre plus ou moins important d'ARNt naturels pouvant être incorporés au niveau du codon stop par le ribosome (même si la reconnaissance ne s'effectuera pas par un appariement complet codon/anticodon).
B. Rôle du contexte nucléotidique du codon stop. Une analyse statistique du contexte nucléotidique des codons stop présents à la fin des gènes chez divers organismes a montré un biais significatif des nucléotides situés en amont et en aval du codon stop. L'influence des deux codons situés en amont du codon stop a lieu principalement au travers des acides aminés incorporés. Ces acides aminés interagissent avec le tunnel de sortie du ribosome et modifient probablement ainsi l'affinité du ribosome pour les facteurs de terminaison.
Dans tous les organismes étudiés le nucléotide situé immédiatement après le codon stop présente un très fort biais de représentation. Ce résultat a entraîné certains scientifiques à émettre l'hypothèse que le signal d'arrêt de la traduction ne serait pas un triplet mais un quadruplet (7). Cependant, même si de nombreuses expériences ont démontré l'importance de ce 4ème nucléotide pour l'efficacité de terminaison de la traduction, aucune preuve n'a jamais été apportée quant à un signal composé de 4 nucléotides au lieu de trois. De plus, aucune règle générale n'a pu être déduite des différentes études réalisées car la nature du nucléotide dépend du codon stop ainsi que de l'organisme étudié. D'autres travaux ont mis en évidence que le contexte influençant l'efficacité de terminaison de la traduction était encore plus étendu (6-9 nucléotides) que ce qui avait été préalablement estimé (8, 9). Plusieurs hypothèses ont été avancées pour expliquer le rôle du contexte nucléotidique situé en aval du codon stop (appariement avec l'ARN ribosomique, interaction avec des protéines du ribosome), mais aucune preuve expérimentale n'a encore été apportée pour confirmer ou infirmer ces hypothèses.
Que se passe-t-il lorsque le ribosome ne s'arrête pas au codon stop?
Nous venons de voir que la présence d'un codon stop ne signifie pas systématiquement l'arrêt de la synthèse protéique. En fonction de la nature du codon stop et du contexte nucléotidique il est possible d'atteindre plus de 30% de passage du codon stop (suppression, ou translecture) chez la levurelevure Saccharomyces cerevisiaeSaccharomyces cerevisiae. Après avoir franchi le codon stop, le ribosome continue la traduction (dans la même phase) jusqu'au codon stop suivant. Cela aboutit à la synthèse d'une protéine possédant une extension carboxy-terminale. Cette extension peut avoir plusieurs conséquences : modifier la stabilité de la protéine, modifier son activité, ou la localisation de la protéine dans la cellule. Il est aussi possible que cette extension possède une nouvelle activité enzymatiqueenzymatique. Ce dernier cas est fréquemment retrouvé dans l'expression de gènes viraux qui utilisent un mécanisme de translecture pour exprimer deux protéines à partir du même ARNm.
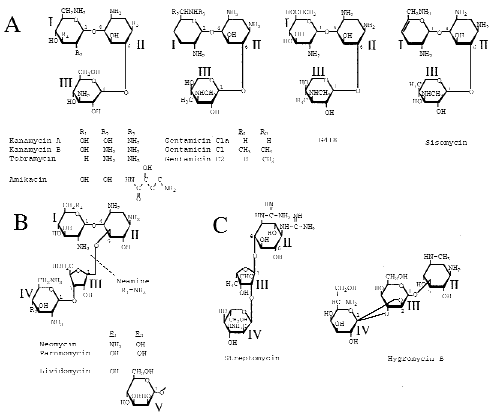
C. Les droguesdrogues. Le ribosome est une cible privilégiée pour de nombreuses drogues notamment les antibiotiquesantibiotiques. Ceux-ci, en inhibant la synthèse protéique des bactéries, permettent de lutter efficacement contre les infections. Le ribosome eucaryote est structuralement bien différent du ribosome procaryote. Ceci implique donc que les molécules actives sur le ribosome procaryote n'auront pas le même effet (voir pas d'effet du tout) sur le ribosome eucaryote. Certains antibiotiques ont la particularité de modifier la fidélité de la traduction, en rendant le ribosome moins fidèle. Nous allons maintenant présenter dans le chapitre suivant une classe particulière d'antibiotiques : les aminoglycosidesaminoglycosides.