au sommaire
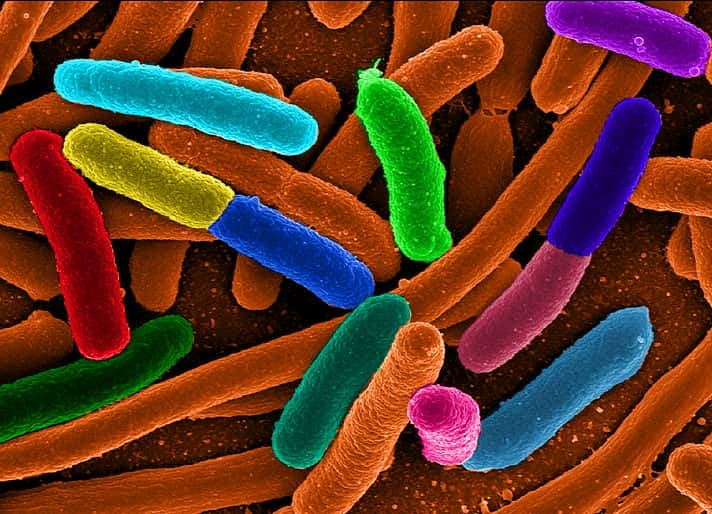
La célèbre bactérie Escherichia coli, modèle si souvent utilisé par la recherche, a fait progresser la génétique d'un cran supplémentaire en devenant le premier organisme entier génétiquement recodé. Après les OGM, voici peut-être venue l'heure des OGR. © Mattosaurus, Wikipédia, DP
La vie sur Terre a toujours évolué selon le même modèle : des êtres disposant d'acides nucléiques (ARN et ADN) portant des informations nécessaires à la synthèse de protéines, ayant des fonctions biologiques indispensables à la survie des cellules. À quelques exceptions près, ce code génétique est universel et lu de la même façon par une bactérie, une cellule de chêne ou d'Homme. Une propriété très intéressante exploitée par les biotechnologiesbiotechnologies, qui permet par exemple de synthétiser de l'insuline humaine grâce à des levureslevures génétiquement modifiées, afin d'aider les personnes diabétiques.
Mais l'utilisation des OGM est limitée par d'autres contraintes, deux principalement. Les bactéries fabriquant des protéines d'intérêt sont souvent victimes de virus, ce qui limite la production. D'autre part, par transfert horizontal, ces gènesgènes particuliers peuvent circuler, intégrer et ainsi contaminer le génomegénome d'autres organismes, et se répandre dans la nature.
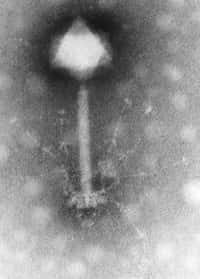
Cependant, un groupe de chercheurs états-uniens codirigé par Farren Isaacs (université Yale) et George Church (université Harvard) vient de proposer une solution outrepassant les limites mentionnées plus haut : les OGR, ou organismes génétiquement recodés. Les scientifiques viennent de publier dans Science leurs résultats concernant une bactérie Escherichia coliEscherichia coli dont le génome a été altéré pour produire des protéines qui ne sont pas fabriquées dans la nature et qui lui permettent de résister à l'infection par le bactériophagebactériophage T7.
Petites bases de génétique
Reprenons la base. L'ADN se compose d'un alphabet de quatre lettres correspondant à quatre bases azotéesbases azotées différentes : A, C, G et T (cette dernière devenant U dans l'ARNARN). Regroupées par trois, elles forment un total de 64 codonscodons, chacun codant pour l'un des 20 acides aminésacides aminés existant naturellement et constituant la base des protéines. Grâce à la redondance du code génétiquecode génétique, différents triplets peuvent aboutir à une même brique peptidique.
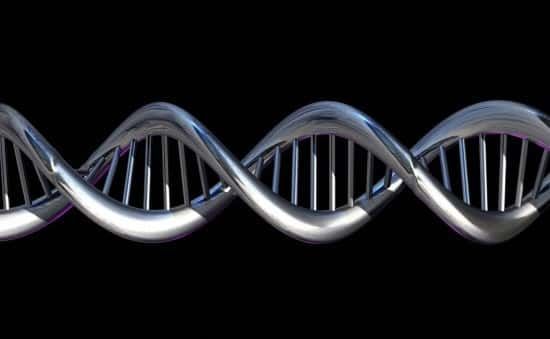
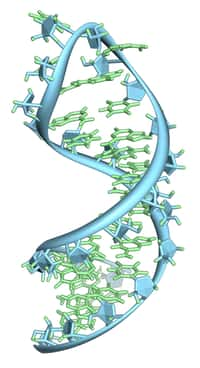
L'ADN est une longue molécule dans laquelle les bases azotées s'enchaînent et composent le code génétique, dont une partie est traduite en protéine selon des règles précises. © Spooky Pooka, Wellcome Images, cc by nc nd 2.0
Les acides aminés viennent se lier les uns aux autres dans un ordre bien précis, jusqu'à ce que la machinerie cellulaire aboutisse à un codon stop, le signal qui permet d'arrêter la traduction et de libérer la protéine ainsi formée. Ceux-ci sont au nombre de trois dans l'ARN : UAG, UAA et UGA.
Chez les bactéries, lorsque la machinerie cellulaire tombe sur l'un de ces triplets, des facteurs de terminaison interviennent afin de mettre fin au processus de traduction. Le codon UAG est reconnu par RF1, UGA par RF2, et UAA est interprété par ces deux facteurs.
Des codons stop qui prennent du sens
Une fois ces données en tête, le reste sera plus simple à comprendre. Car chez E. coli, les biologistes ont systématiquement remplacé tous les codons stop UAG (321 en tout) par des UAA, à l'aide de techniques de biologie moléculairebiologie moléculaire modernes. Cette manipulation n'altère en rien le produit final de l'expression des gènes.
Ensuite, ils ont fait de ces bactéries génétiquement modifiées des mutants incapables d'exprimer RF1, ce qui, une fois encore, ne change en rien la nature des protéines synthétisées par E. coli. C'est alors que les chercheurs ont réinjecté des codons UAG sur certains gènes de la bactérie et l'ont modifiée pour qu'elle les interprète comme des triplets codant pour un nouvel acide aminé, apporté dans le milieu : c'est un recodage génétique.
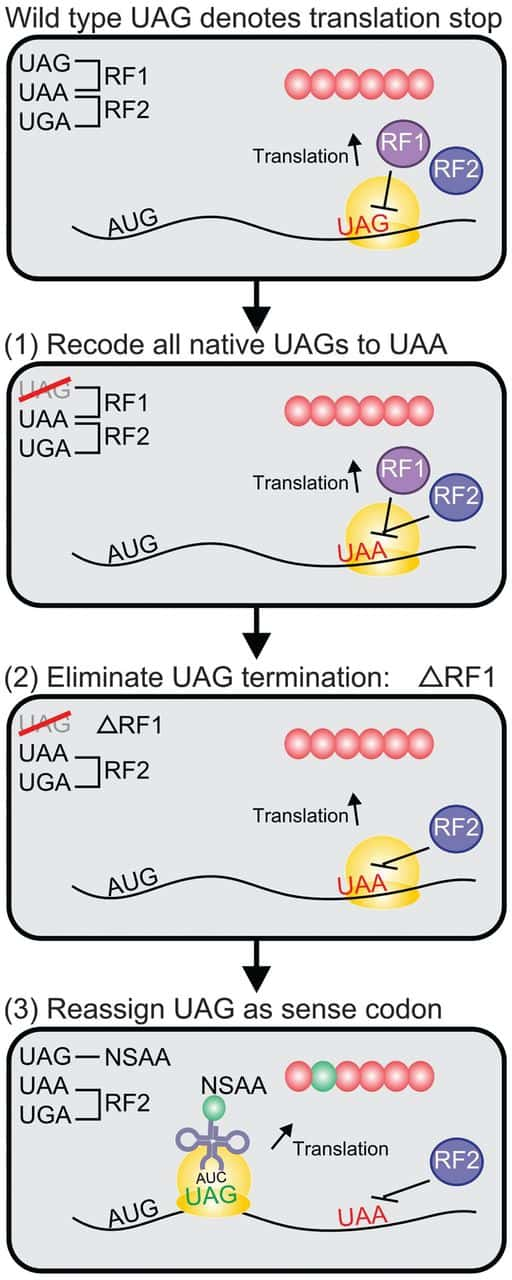
Ce schéma reprend les différentes étapes de l’expérience des chercheurs états-uniens. À partir d'un profil sauvage, le codon UAG est retiré de tous les gènes, et remplacé par UAA (1), qui est interprété comme un codon stop par RF1 et RF2. Puis on crée des mutants qui ne synthétisent plus RF1 (2). Ainsi, on peut réintroduire dans la partie codante du gène un codon UAG (3), qui va alors coder pour un acide aminé non conventionnel. © Marc Lajoie et al., Science
Les OGR, l’avenir des OGM ?
Pour tester l'intérêt de leur OGR, les auteurs ont soumis ces E. coli à deux de ses pires ennemis : les bactériophages T4T4 et T7. Si le premier n'a pas été affecté par les modifications du génome, le second s'est montré plus à la peine lorsqu'il s'agissait d'infester sa proie privilégiée. Le recodage renforce donc la résistancerésistance bactérienne aux virusvirus.
D'autre part, avec l'ajout de ces acides aminés que la vie n'avait pas prévus, le transfert horizontal n'aboutira pas à l'expression de protéines fonctionnelles, ce qui limite très fortement les risques de contaminationcontamination et d'expansion à d'autres organismes.
Enfin, les scientifiques espèrent même qu'à terme, ces nouveaux peptidespeptides pourraient constituer de nouveaux médicaments exploitables par l'industrie pharmaceutique pour proposer des solutions contre des maladies contre lesquelles il n'existe pas de traitement, ou bien lorsque ceux-ci ne sont pas pleinement efficaces. Certains imaginent même l'émergenceémergence de nouveaux matériaux pouvant intégrer certaines nanostructures. De quoi augurer de belles innovations grâce à ces OGR.