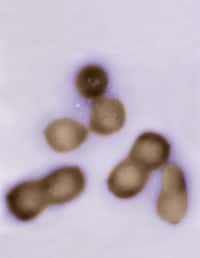
Ces robots, fabriqués à partir de brins d’ADN, se créent et se régénèrent comme les cellules d’un organisme vivant. Grâce à l’ingénieux système mis au point par les chercheurs, ils sont aussi capables de se déplacer et peut-être d’auto-évoluer en tirant parti des mutations naturelles de l’ADN.
au sommaire
Les chercheurs s'inspirent depuis longtemps de la nature pour créer des robots ressemblant à des organismes vivants. On a ainsi pu voir des robots-méduses, un « Bat bot » imitant le vol de la chauve-sourischauve-souris, un drone colibri, des robots miniatures cafards et serpents pour inspecter et réparer les moteurs d'avions ou même des microrobots élastiques inspirés des bactéries. Depuis les années 2000, des nanorobots moléculaires fabriqués à partir de brins d'ADN ont aussi vu le jour. En 2017, des chercheurs du Caltech (California institute of technology) ont ainsi construit des robots ADN capables de se déplacer et de transporter des « colis » de moléculesmolécules, qui pourraient par exemple servir à apporter des médicaments vers des cellules ciblées à l’intérieur du corps.
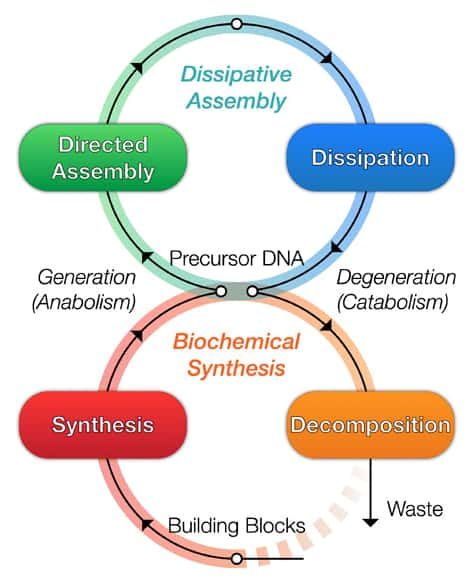
Mais toutes ces approches n'ont pas grand-chose à voir avec un véritable organisme vivant, dont les cellules se renouvellent et s'assemblent de façon autonome. Dan Luo et son associé de recherche Shogo HamadaHamada de l'université de Cornell affirment aujourd'hui avoir créé le tout premier microrobot en matériaumatériau biosynthétique et doté d'un métabolismemétabolisme artificiel. « Notre matériau n'est pas à proprement parler vivant mais il s'approche d'un comportement biologique d'une façon plus réaliste que jamais réalisée auparavant », assure Dan Luo. Pour leur expérience, publiée le 10 avril dans la revue Science Robotics, les chercheurs ont créé un système appelé Dash (DNA-based Assembly and Synthesis of Hierarchical), capable de créer de l'ADN pouvant s'assembler, s'organiser et se régénérer, « trois caractéristiques clés de la vie », appuie Dan Luo.
Se déplacer comme une moule dans la vase
Les chercheurs sont partis d'un bloc de 55 nucléotidesnucléotides (les molécules de base de l'ADN) qu'ils ont multiplié des centaines de milliers de fois pour créer des chaînes d’ADN de quelques millimètres de long. Ils ont ensuite élaboré deux microfluides, l'un ayant la capacité d'assembler l'ADN et l'autre de le dégrader. Ils ont alors injecté les chaînes de nucléotides dans le mélange, la « queue » de l'ADN étant dégradée au fur et à mesure que la « tête » est créée.
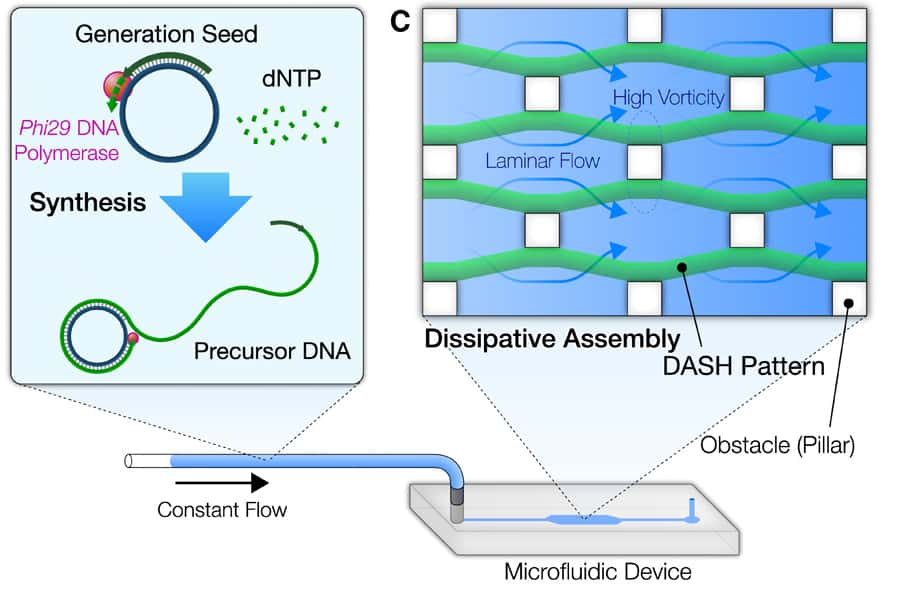
« La biosynthèse et la dégradation sont des éléments clés du métabolisme, à l'instar d'un organisme vivant où de nouvelles cellules se créent en permanence pour remplacer les cellules mortes », explique Dan Luo. Cette dynamique permet aussi au microrobot de se déplacer à contre-courant dans le microfluide au milieu d'obstacles, un peu à la manière des moules de vase qui progressent au fond de l'eau par des étirements et des contractions. Les chercheurs étudient à présent un moyen de faire reconnaître des stimuli au robot, pour aller chercher de la nourriture ou éviter de la lumièrelumière par exemple.
Des robots évolutifs en fonction des mutations de l’ADN
Le robot n'a résisté pour l'instant qu'à deux cycles de synthèse et de dégradation avant son expiration. Mais sa longévité pourra certainement être prolongée et on pourrait même générer des machines entièrement nouvelles au fur et à mesure des réplicationsréplications, en tirant parti des mutations naturelles de l'ADN. « Un peu comme pour l'évolution naturelle », illustre Dan Luo.
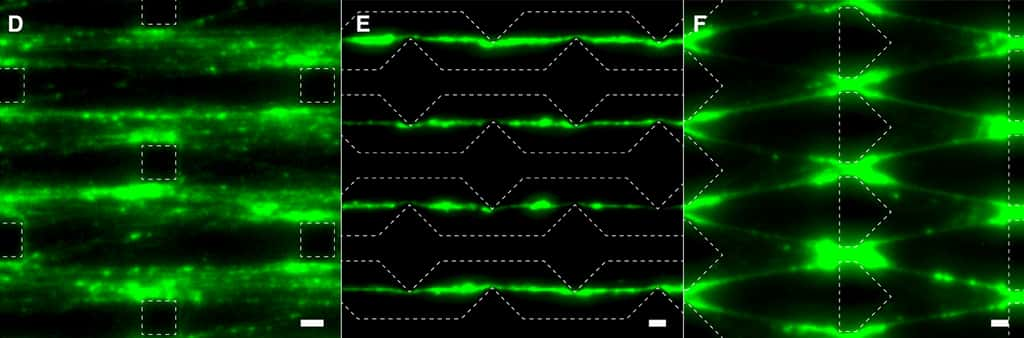
La conception est encore très primitive et on est loin d'avoir de véritables amas de cellules capables de s'organiser comme celles d'un organisme vivant. L'idée des chercheurs est pour l'instant d'exploiter ces biomatériaux comme des capteurscapteurs pour détecter de l'ADN ou pour fabriquer des protéinesprotéines sans cellule vivante. « Le métabolisme artificiel ouvre la voie à une nouvelle frontière en robotique », assure Shogo Hamada. « La vie a commencé il y a des milliards d'années à partir de quelques molécules. C'est peut-être la même chose avec notre matériau. »
Le premier organisme semi-synthétique stable
Article de Nathalie MayerNathalie Mayer publié le 28/01/2017
Où donc arrêtera-t-on la manipulation génétiquegénétique ? La question peut à nouveau se poser aujourd'hui, après l'annonce faite par des chercheurs d'un institut américain. Ils sont parvenus à modifier le patrimoine génétique de bactériesbactéries en l'enrichissant de deux nouvelles bases azotéesbases azotées totalement artificielles. Mieux encore, ils ont réussi à maintenir ces bactéries en vie et à faire se reproduire ce nouveau génomegénome.
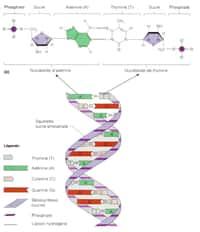
AdénineAdénine (A), thyminethymine (T), cytosinecytosine (C) et guanineguanine (G) : ainsi se nomment les bases qui composent l'ADN. Des arrangements différents et voici que naissent des bactéries ou des papillons, des pingouins ou même des Hommes. Si une telle biodiversitébiodiversité peut être obtenue à partir de quatre bases seulement, que pourrions-nous espérer d'un code à six bases ? C'est la question à laquelle espèrent répondre des chercheurs américains du Scripps Research Institute (TSRI). Pour ce faire, rien de tel qu'un organisme semi-synthétique.
Une première étape avait été franchie en 2014. Les chercheurs américains avaient alors développé deux nouvelles bases, baptisées X et Y. Ils étaient parvenus à les intégrer au cœur de l'ADN de bactéries Escherichia coliEscherichia coli. Problème : la santé des bactéries ainsi génétiquement modifiée -- des bactéries semi-synthétiques -- s'était avérée fragile. Et au fil des divisions cellulaires -- qui assurent la survie de l'espèce unicellulaire --, les bases synthétiques étaient progressivement expulsées des brins d'ADN qui, rapidement, retrouvaient alors leur structure naturelle.
Non contents de ces premiers résultats, bien qu'encourageants, les chercheurs du TSRI ont persévéré. Il y a quelques jours, ils ont annoncé avoir trouvé la clé du problème. Ainsi, sont-ils parvenus à donner naissance à un tout premier organisme semi-synthétique qu'ils qualifient eux-mêmes de « réellement vivant », car disposant d'un génome enfin stabilisé. La preuve ? Les organismes inclus dans l'étude sont demeurés semi-synthétiques, même après 60 divisions cellulaires, une quasi-infinité dans ce cas.

Un correcteur d’orthographe au cœur des cellules
Pour arriver à leurs fins, les chercheurs américains ont d'abord travaillé à optimiser le transporteur de nucléotides déjà utilisé en 2014. Indispensable à la duplication des bases synthétiques, celui-ci affichait en effet une fâcheuse tendance à affaiblir les bactéries cobayes. Ils ont également choisi de modifier la chimiechimie de la base Y pour en faire une base plus facilement reconnaissable par les enzymesenzymes chargées de la synthèse de l'ADN lors du processus de duplication.
Enfin, les chercheurs du TSRI se sont appuyés sur un outil dont la popularité en génie génétique ne cesse de croître : une sorte de ciseaux génétiques baptisés CRISPR-Cas9CRISPR-Cas9. Les scientifiques le prononcent « crispère » et l'utilise pour cibler une zone spécifique de l'ADN, la couper et y insérer ensuite une autre séquence. Mais les chercheurs américains lui ont rendu son rôle primordial. Au cœur de la bactérie en effet, CRISPR-Cas9 joue un peu un rôle de catalyseurcatalyseur de réponse immunitaireréponse immunitaire. Lorsque se présente un ennemi, virus ou autre, CRISPR-Cas9 prélève un fragment de son ADN et l'intègre à son propre génome. Ainsi, en cas de nouvelle attaque, il sera plus simple et rapide de diriger une enzyme contre cet ennemi reconnu. Dans le cas présent, CRISPR-Cas9 a donc été transformé en véritable système de vérification orthographique capable d'identifier les ADN naturels comme indésirable, favorisant ainsi la survie des ADN semi-synthétiques.
Pour tempérer un peu les enthousiasmes naissants -- mais aussi les craintes qui pourraient accompagner de telles manipulations de l'essence même de la vie --, les auteurs de l'étude rappellent toutefois que ces travaux, menés sur un organisme unicellulaire, n'ont pas encore pu être étendus au-delà. Quant aux éventuelles applicationsapplications qui pourraient en résulter, il faudra encore bien des avancées pour seulement commencer à les entrevoir. Il faudra notamment déterminer la façon dont ce nouveau code génétiquecode génétique peut être traduit en protéines avec l'espoir de fabriquer sur mesure, de nouvelles molécules thérapeutiques.
Un nouvel ADN à six lettres au lieu de quatre créé au laboratoire
Article de Janlou Chaput, paru le 12/05/2014
Des biologistes viennent de réussir une prouesse que certains pensaient impossible. Ils ont créé une bactérie dotée d'un ADN semi-synthétique, composé non pas de quatre lettres comme d'ordinaire, mais de six. L'être unicellulaire s'est divisé et ses descendants en ont également profité, laissant entrevoir de nombreuses avancées dans le domaine des biotechnologies.
L'ADN, c'est comme le baseball : tout repose sur quatre bases. Néanmoins, l'analogieanalogie entre la molécule de la vie et le sport s'arrête ici. Tout curieux de la biologie sait que l'acide désoxyribonucléiqueacide désoxyribonucléique se compose d'un sucresucre (le désoxyribosedésoxyribose) et de quatre molécules qui s'apparient, les nucléotides, appelés aussi bases azotées. Ces bases sont surnommées A (adénine), C (cytosine), G (guanine) et T (thymine). Elles sont identiques d'une espèceespèce à l'autre, de la bactérie parasite au poissonpoisson-chat en passant par le muguetmuguet ou le joueur de baseball, et se lient toujours de la même façon : lorsqu'un brin porteporte un A, l'autre porte un T (ou inversement), alors que C et G jouissent d'une grande affinité et vont toujours par paire.
Cependant, un alphabet à quatre lettres connaît forcément des limites. C'est pourquoi les scientifiques tentent depuis des décennies d'insérer de nouvelles lettres pour augmenter les potentialités de l'ADN. Mais la tâche est ardue, car celles-ci doivent impérativement être compatibles avec la machinerie enzymatiqueenzymatique à l'origine de la duplication de la longue molécule.
En 1989, le biologiste Steven Benner réussissait à créer des cytosines et des guanines modifiées, intégrées dans l'ADN. Mais aujourd'hui, l'équipe de Floyd Romesberg, du Scripps Research Institute de La Jolla (Californie, États-Unis), est allée bien plus loin. Après 15 années de recherche, ces scientifiques ont créé deux nouvelles bases azotées, bien différentes de celles déjà existantes, qu'ils ont pu intégrer dans un génome bactérien et qui a été transmis à la descendance. L'essentiel à retenir est livré dans la revue Nature.
A, C, T, G… X et Y
Par le passé, plusieurs molécules absentes chez les êtres vivants ont pu être appariées in vitroin vitro. Dans une étude de 2008, les auteurs avaient déjà travaillé sur 60 candidats potentiels avant de les scanner afin d'évaluer les 3.600 possibilités. De ce travail, deux composés semblaient sortir du lot : d55ICS et dNaM. Leur avantage : leur compatibilité avec les enzymes qui recopient et traduisent l’ADN.

Des tests sont effectués dans des tubes à essaitubes à essai. Avec succès. Les deux molécules, surnommées X et Y, se comportent comme espéré, se sont reproduites et ont été transcrites en ARNARN. Il fallait alors passer à l'étape suivante : dans un organisme vivant.
Comme souvent, c'est la bactérie Escherichia coli qui joue les cobayes. Il a fallu la modifier génétiquement en ajoutant un gènegène d'une diatomée (une alguealgue unicellulaire), qui rend la membrane plus poreuse et laisse passer les molécules. Ensuite, les scientifiques ont créé des boucles d'ADN, nommées plasmidesplasmides, contenant un seul exemplaire de la paire XY. Ces plasmides ont été intégrés dans le microbemicrobe grâce à sa nouvelle aptitude à laisser pénétrer les grosses molécules.
Des bactéries qui maîtrisent un ADN à l’alphabet amélioré
Les enzymes des E. coli ont effectivement reproduit le plasmide, en récupérant les deux bases azotées inédites dans leur environnement. Lorsque celui-ci s'est vidé de ses ressources, les bactéries ont pu reprendre leurs habitudes usuelles et recourir à une molécule d'ADN avec les quatre lettres originelles. Un garde-fougarde-fou pour les auteurs, qui permet de contrôler les populations génétiquement modifiées sans risque de les voir se répandre dans la nature.
Un résultat extraordinaire applaudi par toute la communauté scientifique, dont certains membres doutaient de la possibilité d'une telle prouesse. Il reste cependant beaucoup de travail. La prochaine étape : s'assurer que cette technique permet la formation de nouvelles protéines inédites dans la nature et difficiles à synthétiser en laboratoire. Ainsi, en disposant de propriétés nouvelles, il est possible d'imaginer des molécules inconnues et utiles.
Sera-t-il possible un jour de créer de toutes pièces un organisme génétiquement modifié de telle sorte que son ADN serait complètement non naturel ? Cela est impossible à concevoir avec les moyens actuels, mais nous pouvons malgré tout nous octroyer le droit de rêver...
Ce qu’il faut
retenir
- Des chercheurs ont mis au point un biomatériau en ADN qui se crée et se dégrade au fil des cycles.
- Ce mécanisme lui permet aussi de se déplacer au sein d’un micro fluide par étirement et contraction.
- Ces robots pourraient servir de biocapteurs et « d’usines à protéines » sans avoir recours à des cellules vivantes.