au sommaire
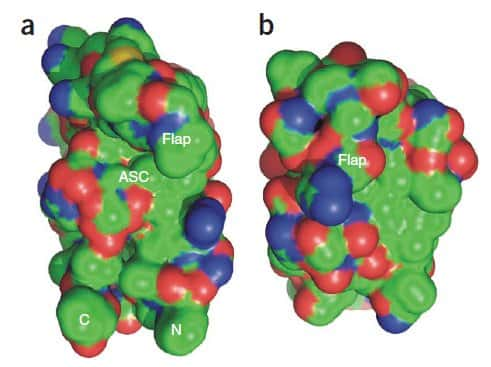
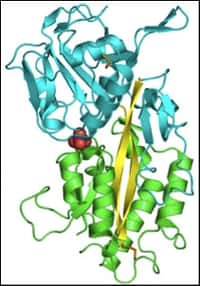
À gauche, la protéase du virus du Sida, le HIV. À droite, celle du M-PMV déterminée grâce aux joueurs de Foldit. Les chercheurs y voient quelque chose de très intéressant : un volet (flap) vient obturer la cavité qui constitue le site actif (ASC). Si l'on parvient à empêcher l'ouverture de ce volet, la protéine est inactive et le virus ne se reproduit pas. © Firas Khatib et al.
Ils sont une quinzaine et appartiennent à un groupe de joueurs en ligne baptisé Contenders (« les compétiteurs »). Leur score déjà élevé les a fait choisir pour un nouveau défi. Le jeu ? Foldit, une initiative de David Baker et son équipe pour l'expérience Casp (Critical Assessment of protein Structure Prediction, Évaluation pour une prédiction de la forme des protéinesprotéines), qui avait aussi lancé le projet Rosetta@Home. Le but est de proposer à des bénévoles de travailler pour la science. De la même manière que Seti@Home demande du temps de calcul sur les ordinateurs personnels des volontaires, Foldit (« Plie-la ») cherche la puissance... cérébrale.
Aujourd'hui, le jeu comporte environ 100.000 joueurs et pas mal de fanas parmi eux. Le défi relevé par l'équipe Contenders était de découvrir la forme exacte de la protéaseprotéase rétrovirale, une enzymeenzyme qui permet la multiplication du M-PMV, pour Mason-PfizerPfizer monkey virus, un rétrovirusrétrovirus responsable de l'une des formes de SidaSida chez le macaque Rhésusmacaque Rhésus. Les biochimistesbiochimistes calaient sur ce problème depuis plus de dix ans ! Résultat de trois semaines de compétition, après vérification par les chercheurs : une publication dans Nature Structural and Molecular Biology. La forme semble la bonne et ouvre une piste pour une nouvelle famille d'antivirauxantiviraux qui pourraient être efficaces contre le HIV du Sida humain.

Une image rare : parmi les auteurs d'un article scientifique figure celui d'un groupe de joueurs d'un jeu en ligne... (Image de la publication dans Nature Structural and Molecular Biology)
« L’ingéniosité des joueurs est une force formidable »
Les problèmes à résoudre dans Foldit concernent le pliage des protéines, une activité qui, elle, n'est pas du tout un jeu. Gigantesques moléculesmolécules, les protéines sont constituées de plusieurs centaines d'acides aminésacides aminés et chaque liaison entre deux de ces maillons forme un axe de rotation. Les possibilités de pliage sont donc énormes, conduisant à des formes finales très différentes. Or, la forme d'une protéine est absolument déterminante pour son fonctionnement, qui est celui d'une enzyme, c'est-à-dire d'un catalyseurcatalyseur hyperspécifique d'une certaine réaction chimiqueréaction chimique. On sait déterminer la succession des acides aminés (par exemple en analysant le gènegène qui lui correspond) mais à partir de là on a beaucoup de difficultés à prédire sa forme.
Il existe des logiciels pour faire ce travail mais les temps de calcul sont très longs. Le projet Foldit repose sur l'idée que l'intelligenceintelligence humaine, à coup d'intuitions ou de réflexion, peut faire beaucoup mieux, surtout avec de très nombreux participants, même s'ils n'ont aucune connaissance en biochimiebiochimie. Seth Cooper et les autres concepteurs du jeu ont réalisé une superbe applicationapplication 3D. Les joueurs doivent résoudre des « puzzles » (c'est le terme) sur des thèmes imposés, pour trouver des conformationsconformations de protéines viables et qui leur semblent les meilleures. Le score est calculé en fonction de l'état d'énergieénergie du modèle proposé, le meilleur étant celui qui en représente le moins et qui est donc le plus stable (c'est l'une des approches utilisées pour déterminer la forme d'une protéine). Le joueur peut réagir et chercher un moyen d'augmenter son score. Selon le magazine Wired, l'un des bons joueurs, Cheese de son pseudo, a treize ans... Il serait un jour entré dans une compétition serrée avec un Français, Laurent de Jerphanion, pour la résolution du puzzle T0461.
Les résultats sont ensuite analysés par des vrais biochimistes, pour être éventuellement testés par des expériences de cristallographiecristallographie. Il est vite apparu lors de l'expérience Casp9 que l'équipe travaillant sur la protéase du M-PMV marquait des progrès constants. « Remarquablement, malgré l'échec complet de toutes les approches antérieures, plusieurs solutions du groupe Contenders sont apparues suffisamment bonnes pour permettre une rapide compréhension de la structure cristalline. » En découvrant cette forme, les chercheurs imaginent déjà un moyen pour inactiver le site actifsite actif de la protéase du HIV, celui du Sida des humains. « L'ingéniosité des joueurs est une force formidable » concluent les biochimistes.